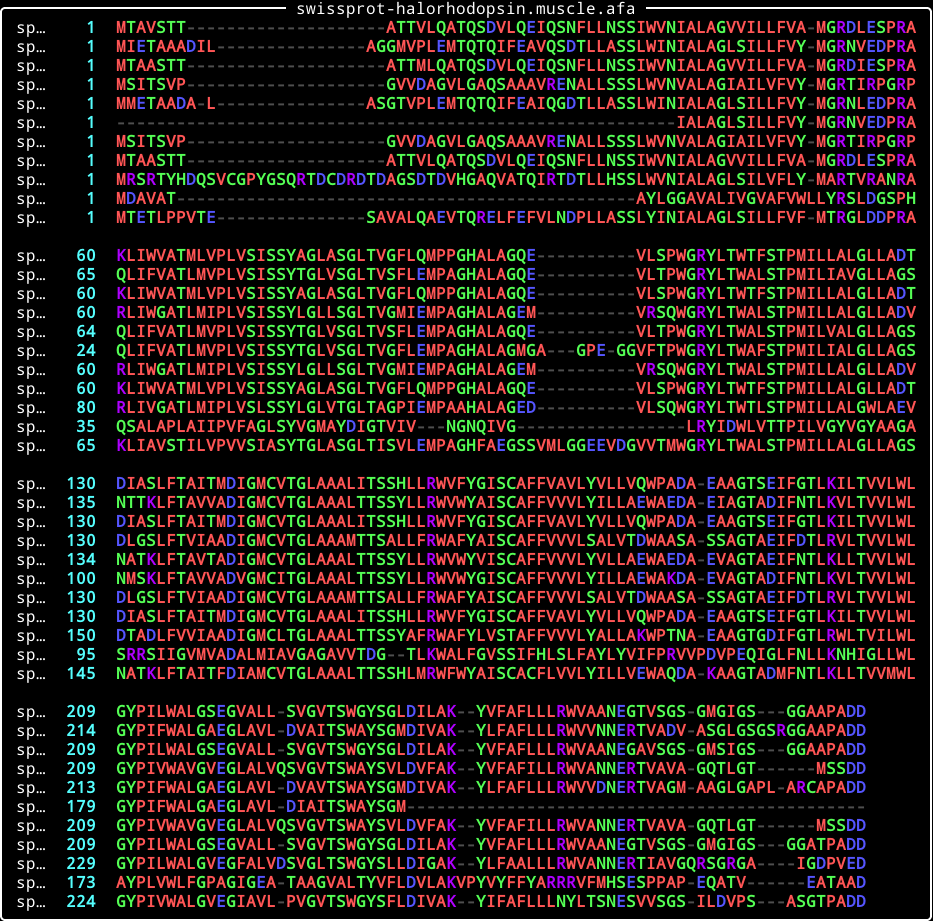
在终端中查看多个序列比对的可渲染丰富的组件。
项目描述
rich-msa 
一个简单的模块,使用rich在终端中渲染彩色多个序列比对。
🔧 安装
直接从PyPi安装rich-msa包,它托管通用的wheel,可以使用pip安装
$ pip install rich-msa
💡 示例
使用Biopython从对齐的FASTA文件中加载MSA,并将其渲染到终端
import Bio.AlignIO
import rich
from rich_msa import RichAlignment
msa = Bio.AlignIO.read("tests/data/swissprot-halorhodopsin.muscle.afa", "fasta")
viewer = RichAlignment(
names=[record.id for record in msa],
sequences=[str(record.seq) for record in msa],
)
panel = rich.panel.Panel(viewer, title="swissprot-halorhodopsin.muscle.afa")
rich.print(panel)
您应该得到类似以下图片的输出,根据您的终端宽度进行缩放:
🪛 命令行
如果您已从PyPI安装了rich-msa库,则可以直接使用它来查看任何对齐文件,前提是您已安装Biopython。
$ python -m rich_msa -i tests/data/swissprot-halorhodopsin.muscle.afa
使用-f标志将文件格式从默认的对齐FASTA格式更改为Biopython支持的任何对齐格式。
💭 反馈
⚠️ 问题跟踪器
发现了一个错误?有一个增强请求吗?如果您需要报告或询问某些问题,请访问GitHub问题跟踪器。如果您正在报告错误,请尽可能多地提供有关问题的信息,并尝试在简单、易于复现的情况下重现相同的错误。
🏗️ 贡献
欢迎贡献!有关详细信息,请参阅CONTRIBUTING.md。
⚖️ 许可证
此库受MIT许可证的许可。
本项目与原始Rich作者无任何关联、赞助或支持。它是由Martin Larralde在他的欧洲分子生物学实验室的Zeller团队的博士项目中开发的。
项目详情
下载文件
下载适用于您平台的文件。如果您不确定要选择哪个,请了解有关安装软件包的更多信息。
源代码分发
rich-msa-0.1.0.tar.gz (7.2 kB 查看哈希值)
构建分发
rich_msa-0.1.0-py3-none-any.whl (7.4 kB 查看哈希值)
关闭
rich-msa-0.1.0.tar.gz的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 1fd78e16ab4e496f365512eb84a85c3352844e5972ef22d1bf682632482bd664 |
|
| MD5 | 4b7afae4a6ec532be150f675b0210af0 |
|
| BLAKE2b-256 | 858db57eb3386f9f62c00fd5c6c6d31f0a24227b4b86b762d1340e0656755034 |
关闭
rich_msa-0.1.0-py3-none-any.whl的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | bbd7e7a479fff956d3473bb806b66e760f1a10e0569fb703e9e499f1c29ee3c2 |
|
| MD5 | 6bbb2db4525538c39d61345e12e2048c |
|
| BLAKE2b-256 | fdd9a8efa90c881ff4e6f223b7d07db99447d7f1c0c2b8e8998cd8d30c1d8a8e |