OrthoANI算法用于核苷酸同源性度量的实现。
项目描述
OrthoANI 
OrthoANI算法用于核苷酸同源性度量的Python实现。
🗺️ 概述
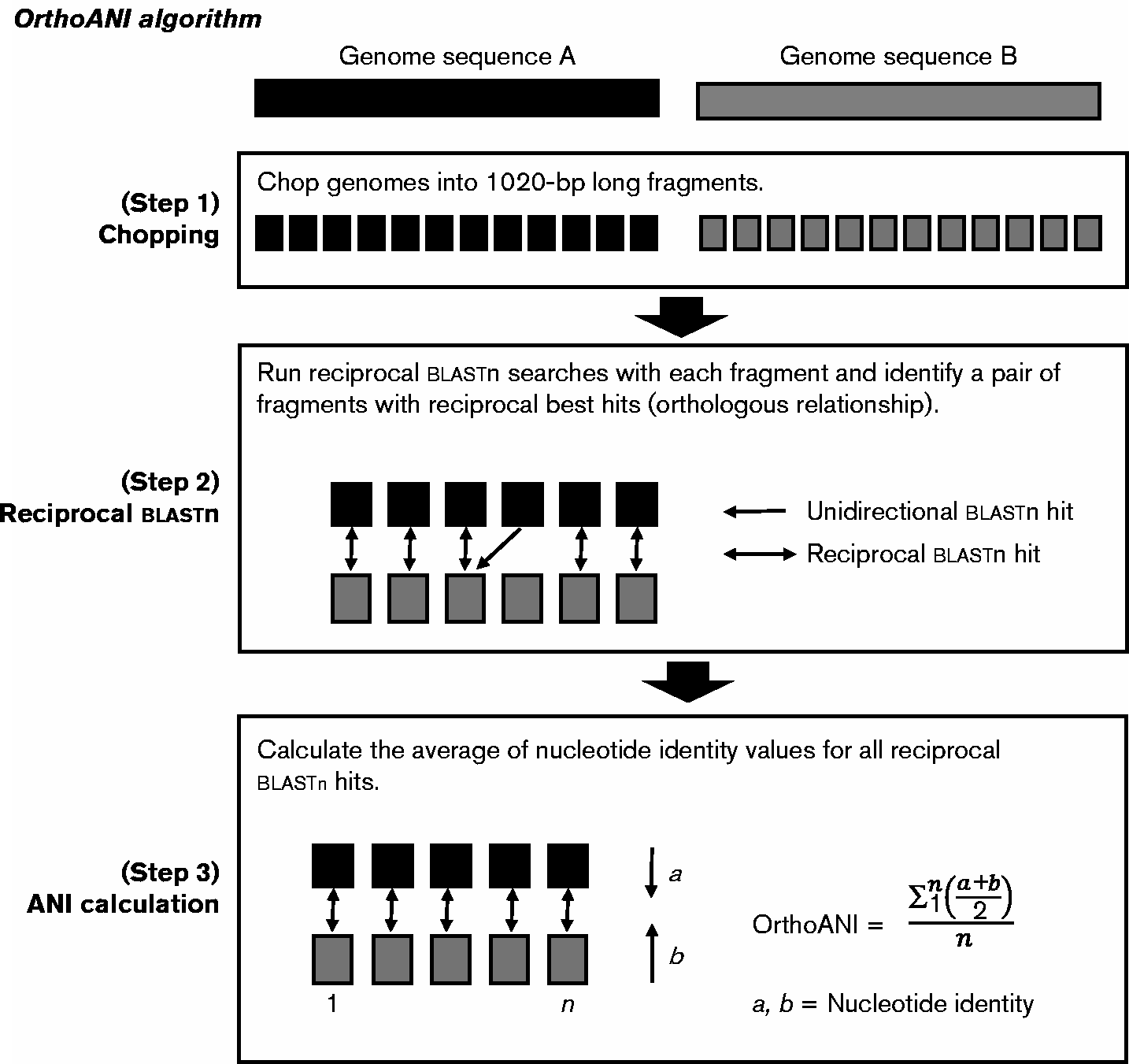
OrthoANI是Lee等人于2016年提出的一种度量,用于改进平均核苷酸同源性计算。它使用BLASTn在两个序列中找到直系同源块,然后只考虑相互直系同源的比对来计算平均同源性。
本项目是对作者在ezbiocloud.net上提供的闭源Java实现的重写。它依赖于Biopython来处理I/O和与BLAST+二进制的交互。
🔧 安装
使用pip安装是最简单的
$ pip install orthoani
orthoani还需要您的机器上安装BLAST+二进制文件,并使其在您的$PATH中可用。
💡 示例
使用Biopython加载两个FASTA文件,然后使用orthoani.orthoani计算它们之间的OrthoANI指标
import orthoani
from Bio.SeqIO import read
genome_1 = read("sequence1.fa", "fasta")
genome_2 = read("sequence2.fa", "fasta")
ani = orthoani.orthoani(genome_1, genome_2)
orthoani还可以通过一个简单的命令行界面从CLI使用
$ orthoani -q sequence1.fa -r sequence2.fa
0.5725
🐏 内存
orthoani使用机器临时文件夹来处理BLAST+输入和输出文件,这可以通过tempfile.tempdir进行配置。[https://docs.pythonlang.cn/3/library/tempfile.html#tempfile.tempdir](https://docs.pythonlang.cn/3/library/tempfile.html#tempfile.tempdir)。在某些系统(如ArchLinux)中,这个文件系统可以驻留在内存中,这意味着您的计算机可能难以处理非常大的文件。如果发生这种情况,请尝试将tempfile.tempdir的值更改为位于物理存储上的目录。
📏 精度
由于Java中的作者在子百分比级别对浮点值进行四舍五入,而此库使用完整值,因此此包计算出的值和原始Java实现可能略有差异。
📜 关于
此库根据开源MIT许可证提供。
该项目与原始OrthoANI作者无关,不受赞助或以其他方式支持。它由Martin Larralde在欧洲分子生物学实验室的Zeller团队攻读博士期间开发。
项目详情
下载文件
下载您平台上的文件。如果您不确定选择哪个,请了解更多关于安装包的信息。