模拟随机系统发育树
项目描述
Ngesh,一个用于系统发育树模拟的库
ngesh是一个Python库和命令行工具,用于模拟系统发育树和相关数据(特征、状态、分支长度等)。它旨在对系统发育方法进行基准测试,尤其是在历史语言学和谱系学中。随机系统发育树的生成也称为“系统发育树的模拟方法”、“合成数据生成”或简称为“系统发育树模拟”。
该包的一些亮点包括,使用ngesh
- 任何可哈希的元素都可以作为伪随机数生成器的种子,保证合成树的可重现性(包括跨不同系统)
- 可以根据用户指定的参数生成树,例如出生和死亡比率(死亡比率可以设置为0,从而生成只有出生的树)
- 树将有随机的拓扑结构,如果需要,还有随机的分支长度
- 可以根据现存的叶子数量、进化时间(与出生和死亡参数相关)或两者来约束树
- 可以从出生-死亡树中修剪掉非现存的叶子
- 物种事件默认有两个后代,但后代数量可以从用户定义的泊松过程中随机抽取(允许模拟硬多歧树)
- 可以根据分支长度模拟特征进化,用户可以指定突变和水平基因转移的比率,并为每个特征指定不同的变化速率
- 节点可以接收唯一的标签,可以是顺序标签(如“L01”、“L02”和“L03”),易于发音的随机名称(如“Sume”、“Fekobir”和“Tukok”),或者是近似二名法标准的随机生物名称(如“Sburas wioris”、“Zurbata ceglaces”和“Spellis spusso”)。
- 树是正常ETE3树对象,可以导出为多种格式,如Newick树、ASCII表示、表格文本列表等。
安装
在任何标准Python环境中,可以使用以下命令安装ngesh:
pip install ngesh
pip安装将获取依赖项ete3和numpy,如果需要的话。来自ete3的内置树可视化工具需要PyQt5库,该库默认不安装,但大多数系统中都应该可用。如果需要,可以与包一起安装:
pip install ngesh[gfx]
如何使用
您可以使用命令行中的ngesh命令测试您的安装,该命令每次调用都会返回不同格式的随机小出生-死亡树。
$ ngesh
((Vovrera:0.149348,(Wigag:3.11592,(Pallo:2.68125,Zoei:1.85803)1:1.29704)1:0.204529)1:0.607805,(((Avi:0.347942,Uemi:0.0137646)1:1.41697,(((Kufo:0.817012,
(Gapurem:0.0203582,Hukub:0.0203582)1:0.796654)1:0.395727,Tablo:0.00846148)1:0.484705,(Kaza:0.140656,((Tozea:0.240634,Pebigmom:0.240634)1:1.13579,(Kata:0
.109977,((Fabom:0.04242,Upik:0.04242)1:0.549364,(Amue:0.182635,Lunida:0.182635)1:0.409149)1:0.366701)1:0.417941)1:0.162968)1:0.158051)1:1.47281)1:1.0326
,(Kunizob:0.650455,Madku:0.221172)1:1.22008)1:0.587783);
$ ngesh
((((Povi:0.325601,Udo:0.325601)1:0.0750448,Hiruta:0.400646)1:0.181454,(Voebi:0.0293506,Sodi:0.0293506)1:0.55275)1:0.258834,((Vandemif:0.0160558,(((Dubik
:0.0543122,Fuvu:0.0543122)1:0.36458,Hitfuv:0.418892)1:0.0388987,Pizuna:0.457791)1:0.0535386)1:0.179893,(Uo:0.67132,Zegna:0.163427)1:0.0199021)1:0.149711
);
相同的命令行工具可以使用文本配置文件中提供的参数。在此,我们生成一个可重复的Yule树的Nexus数据(注意种子123),出生比率为0.666,至少有8个带有"human"标签的叶,以及10个存在/不存在字符。
$ cat ngesh_demo.conf
[Config]
labels=human
birth=0.666
death=0.0
output=nexus
min_leaves=8
num_chars=10
$ ngesh -c ngesh_demo.conf --seed 123
#NEXUS
begin data;
dimensions ntax=16 nchar=38;
format datatype=standard missing=? gap=-;
matrix
Abel 10001001011000010000010010010000100000
Azogu 10001001011000010000010010010000100000
Bou 10001001100010100000010010010000000010
Dipu 10001001010001000010000110010000000001
Gezepsem 10001001100010100000010010010000000010
Gupote 10001001010010010000010010010000000100
Hefi 10100100010010010001000001010001000000
Lerzo 10001001010001000010000110010000000001
Magumel 10001001010010010000010010010000000010
Pao 01001010010100001000100010001000100000
Sanigo 10010100010010000100001000100010010000
Tuzizo 10001001100010100000010010010000000010
Wialum 10001001011000010000010010000100100000
Zudal 10001001010010010000010010010000100000
Zukar 10001001011000010000010010000100100000
Zusu 10010100010010000100001000100010001000
;
end;
配置文件中提供的所有参数都可以在命令行中覆盖。
可以使用-o ascii标志获取同一树的文本表示(即使用参数集和相同种子生成的随机树)。
$ ngesh -c ngesh_demo.conf --seed 123 -o ascii
/-Zudal
|
| /-Azogu
| |
| /-| /-Wialum
| | | /-|
| | \-| \-Zukar
| /-| |
| | | \-Abel
| | |
/-| | | /-Dipu
| | | \-|
| | /-| \-Lerzo
| | | |
| | | | /-Bou
| | | | /-|
| | | | /-| \-Gezepsem
| | /-| | | |
/-| | | | \-| \-Tuzizo
| | | | | |
| | \-| | \-Magumel
| | | |
| | | \-Pao
| | |
--| | \-Gupote
| |
| | /-Zusu
| \-|
| \-Sanigo
|
\-Hefi
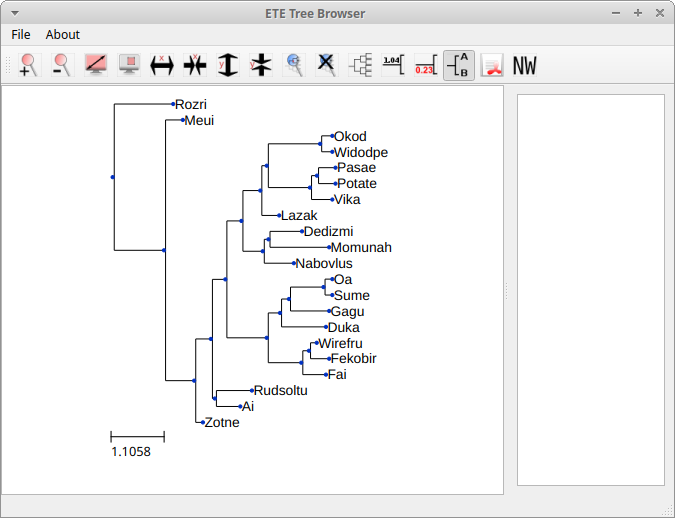
然而,该软件包旨在作为库使用。如果已安装PyQt5,则以下命令将在同一随机树上打开ETE树查看器:
$ ngesh -c ngesh_demo.conf --seed 123 -o gfx
同样,以下代码对于快速演示很有用,每次调用都会弹出查看器并显示随机树。
python3 -c "import ngesh ; ngesh.show_random_tree()"
主要的生成功能是gen_tree()(文档),它返回随机树拓扑,以及add_characters()(文档),它模拟提供的树中的字符进化。由于它们是不同的任务,因此可以只生成随机树或模拟用户提供的树中的字符进化。
下面的代码片段展示了基本的树生成、字符进化和输出流程。
>>> import ngesh
>>> tree = ngesh.gen_tree(1.0, 0.5, max_time=3.0, labels="human")
>>> print(tree)
/-Butobfa
/-|
| | /-Defomze
| \-|
| \-Gegme
--|
| /-Bo
| /-|
| | \-Peoni
\-|
| /-Riuzo
\-|
\-Hoale
>>> tree = ngesh.add_characters(tree, 10, 3.0, 1.0)
>>> print(ngesh.tree2nexus(tree))
#NEXUS
begin data;
dimensions ntax=7 nchar=15;
format datatype=standard missing=? gap=-;
matrix
Hoale 100111101101110
Butobfa 101011101110101
Defomze 101011110110101
Riuzo 100111101101110
Peoni 110011101110110
Bo 110011101110110
Gegme 101011101110101
;
end;
树的Newick表示可以“排序”,解决这些结构比较的问题(请记住,系统发育树就像“悬挂的移动”)。
$ cat tiago.newick
(Ei:0.98,(Mepale:0.39,(Srufo:0.14,Pulet:0.14):0.24):0.58);
$ src/ngesh/newick.py -i tiago.newick
(((Pulet:0.14,Srufo:0.14):0.24,Mepale:0.39):0.58,Ei:0.98);
树生成参数
树生成参数,如命令ngesh -h所示,包括:
birth:树出生率(l)death:树死亡率(mu)max_time:最大进化时间的停止标准min_leaves:最小叶数的停止标准labels:随机标签的文本生成模型(None、"enum"表示简单枚举、"human"表示随机生成的名称和"bio"表示随机生成的物种名称)num_chars:要模拟的字符数量k_mut:字符突变gammak参数th_mut:字符突变gammath参数k_hgt:字符HGT gammak参数th_hgt:字符HGT gammath参数e:字符通用突变e参数
ngesh是如何工作的?
首先,从出生率和死亡率的总和中计算得到一个 event_rate。在每个迭代中,迭代发生在从 event_rate 随机指数分布的时间后,库会选择一个现有的节点进行“事件”:要么是出生要么是死亡,从每个比率的占比中选择。所有其他现有的叶子节点都会用事件时间更新其距离。
随机标签遵循从一组模式中生成随机文本的预期方法,同时注意生成大多数用户都能轻松发音的名字。
对于随机字符生成,它根据与每个分支长度相关的伽马分布参数添加字符。两个可能的事件是突变(假设总是到一个新的字符,即没有平行进化)和水平基因转移。在字符生成过程中不执行任何扰动,例如模拟测序/数据收集中的错误。然而,这些可以通过坏采样模拟函数来模拟。请注意,字符生成仅模拟类似于历史语言学(同源群)的状态,并假设字符独立性(即,没有像词源学中常见的块移动)。虽然我们可能会在将来实现后者,但目前还没有模拟遗传数据的计划。
坏采样在均匀分布中进行模拟,即所有现有的叶子节点都有相同的被移除概率。请注意,如果执行了树拓扑和字符的完整模拟,这项任务必须在字符进化模拟 之后 执行,否则字符将适合采样树而不是原始树。目前没有数据扰动的任何方法,但我们计划在将来实现它们。
与其他软件集成
通过各种导出函数简化了与其他包的集成。例如,可以生成具有所有进化细节和参数的随机树,并生成 Nexus 文件,这些文件可以输入到如 MrBayes 或 BEAST2 等系统发育软件,以检查它们的表现或我们的生成在真实数据方面的好坏。
让我们通过 BEASTling 模拟用于分析 BEAST2 的系统发育数据。我们从一个出生-死亡树(lambda=0.9,mu=0.3)开始,至少有 15 个叶子节点,100 个字符的进化用默认参数建模,并使用字符串种子 "uppsala" 以便可重复;树数据以 "wordlist" 格式导出。
$ cat examples/example_ngesh.conf
[Config]
labels=human
birth=0.9
death=0.3
output=nexus
min_leaves=15
num_chars=100
$ ngesh -c examples/example_ngesh.conf --seed uppsala > examples/example.csv
$ head -n 20 examples/example.csv
Language_ID,Feature_ID,Value
Akup,feature_0,0
Buter,feature_0,0
Dufou,feature_0,0
Emot,feature_0,0
Kiu,feature_0,0
Kovala,feature_0,0
Lusei,feature_0,0
Oso,feature_0,0
Puota,feature_0,0
Relenin,feature_0,976
Sotok,feature_0,0
Tetosur,feature_0,0
Usimi,feature_0,976
Voe,feature_0,0
Vusodur,feature_0,0
Zeba,feature_0,0
Zufe,feature_0,0
Akup,feature_1,1
Buter,feature_1,1
我们现在可以使用一个最小的 BEASTling 配置来生成一个 BEAST2 的 XML 模型。让我们假设我们想测试当数据实际上包括灭绝的物种时,假设 Yule 树时我们的管道表现如何。这里展示的结果并不完美,因为我们使用较短的链长度以使其更快,并且使用了一个与生成假设不同的模型(除了默认参数中水平基因转移的假设对于这个模拟来说太高之外)。
$ cat examples/example_beastling.conf
[admin]
basename=example
[MCMC]
chainlength=500000
[model example]
model=covarion
data=example.csv
$ beastling example_beastling.conf
$ beast example.xml
我们可以像平常一样进行:使用 BEAST2 的 treeannotator(或类似软件)生成一个总结树,我们将其存储在 examples/summary.nex 中,并使用 figtree(或类似软件)绘制结果。
让我们绘制我们的总结树,并将结果与实际拓扑结构进行比较(我们可以使用早期种子重新生成)。
$ ngesh -c examples/example_ngesh.conf --seed uppsala --output newick > examples/example.nw
由于我们为了快速演示而设定的限制,结果并不出色,但它仍然捕捉到了主要信息和子组(如下面的放射状布局所示)——手动数据探索表明,至少一些错误,包括第一次分裂中的群体,是由于水平基因转移引起的。为了分析推断性能,我们需要改进上述参数,并在一系列随机树上进行重复分析,包括研究涉及在这个随机树中的字符变化(包括借用)的日志。
我们可以使用常见的树比较方法来比较树,例如 Robinson–Foulds 指数。所有用于此目的的软件包和编程语言都应该能够读取以 Newick 或 NEXUS 格式导出的树;然而,由于 ngesh 树实际上是 ETE3 树,我们可以直接从 Python 中进行操作。
d = tree1.robinson_foulds(tree_2)
本例中使用和生成的文件可以在 /examples 目录中找到。
“ngesh”是什么意思?
技术上,“ngesh”只是一个独特的名称,来源于苏美尔语中“树”的一个单词 ĝeš。这个名字被选中是因为该库最初计划作为模拟语言进化以及相关工具基准测试的一个更大系统的一部分,该系统以苏美尔语中语言和“随机性”之神 恩基 命名。
按照最被广泛接受的重建,其发音为 /ŋeʃ/。但请不要过于纠结于此,你可以随意将其读作 /n̩.gɛʃ/,就像大多数人所做的那样。
替代方案
有许多工具用于模拟系统发育过程以获取随机系统发育树。最完整的是由 Tanja Stadler 开发的 R 软件包 TreeSim,它包括许多灵活的树模拟函数。在 R 中,还可以使用 ape 软件包中的 rtree() 函数和 geiger 软件包中的 birthdeath.tree() 函数,以及手动在分类图中随机放置类群。
在 Python 中,由 Marc-Rolland Noutahi 在博客文章 如何模拟系统发育树?(第一部分) 中提供的一个片段,其工作方式与 ngesh 类似,并为其提供了最初的灵感。
对于更简单的模拟,ETE 中的 Tree 类的 .populate() 方法可能就足够了。关于此方法的信息可以在 这里 查找。《toytree》和《dendropy》软件包也提供类似的功能。
在编写本文时,有许多在线模拟树的工具可用
- T-Rex(树和网状图重建),位于加拿大魁北克省蒙特利尔大学(UQAM)
- Anvi'o 服务器 可以作为 T-Rex 上述工具的包装在线使用
- phyloT,通过随机抽样物种名称、标识符或蛋白质访问号,可以用于相同的目的
图库
参考文献
-
Bailey, Norman. T. J. (1964). 《随机过程及其在自然科学中的应用》. 约翰·威利父子有限公司。
-
Bouckaert, Remco; Vaughan, Timothy G.; Barido-Sottani, Joëlle; Duchêne, Sebastián; Fourment, Mathieu; Gavryushkina, Alexandra., et al. (2019). "BEAST 2.5: 一个先进的贝叶斯进化分析软件平台". 《PLoS 计算生物学》, 15(4), e1006650. DOI: 10.1371/journal.pcbi.1006650.
-
Foote, Mike; Hunter, John P.; Janis, Christine M.; and Sepkoski J. John Jr. (1999). "生物群起源的进化限制和保存限制:真兽亚目的分化时间". 《科学》283:1310–1314。
-
Harmon, Luke J. (2019). 《系统发育比较方法——从树中学习》. 可在:https://lukejharmon.github.io/pcm/chapter10_birthdeath/. 访问日期:2019-03-31。
-
Huerta-Cepas, Jaime; Serra, Francois; and Bork, Peer (2016). "ETE 3:系统发育基因组数据的重建、分析和可视化". 《分子生物学与进化》。DOI: 10.1093/molbev/msw046。
-
毛里斯,卢克;福克尔,罗伯特;开平,格奥尔格·A.;阿特金森,昆汀·D.(2017)。"BEASTling:使用BEAST 2进行语言系统发育学的软件工具。" PLoS one 12(8),e0180908。DOI:10.1371/journal.pone.0180908。
-
诺塔希,马克-罗兰(2017)。如何模拟一个系统发育树?(第一部分) 可在:https://mrnoutahi.com/2017/12/05/How-to-simulate-a-tree/。访问日期:2019-03-31。
-
罗宾逊,D. R.;福尔德斯,L. R.(1981)。"系统发育树的比较"。数学生物科学 53 (1–2):131–147。DOI:10.1016/0025-5564(81)90043-2。
-
斯塔德勒,塔尼亚(2011)。"使用固定现存物种数量的模拟树"。系统生物学 60.5:676-684。DOI:10.1093/sysbio/syr029。
《ngesh》横幅由蒂亚戈·特雷斯奥迪设计,基于J. 尼德汉姆在尼德汉姆,J. (1895) 用铅笔和彩色水彩画树的 研究。第一系列。伦敦,格拉斯哥,爱丁堡:布莱基父子出版社。 (公有领域,可在archive.org获取)。
社区指南
尽管作者可以直接联系以获得支持,但建议第三方使用GitHub标准功能,例如问题反馈和拉取请求,以贡献、报告问题或寻求支持。
贡献指南,包括行为准则,可在CONTRIBUTING.md文件中找到。
作者和引用
该库由蒂亚戈·特雷斯奥迪开发(tiago.tresoldi@lingfil.uu.se)。该库是在文本文化进化项目背景下开发的,并由瑞典银行纪念基金会资助(资助协议ID:MXM19-1087:1)。
在开发的早期阶段,作者获得了欧洲研究委员会(ERC)的资助,该资助来自欧盟的《地平线2020》研究和创新计划(资助协议号ERC Grant #715618,计算机辅助语言比较)。
如果您使用ngesh,请按照以下方式引用:
特雷斯奥迪,T.(2021)。Ngesh:用于合成系统发育数据的Python库。开源软件杂志,6(66),3173,https://doi.org/10.21105/joss.03173
在BibTeX中
@article{Tresoldi2021ngesh,
doi = {10.21105/joss.03173},
url = {https://doi.org/10.21105/joss.03173},
year = {2021},
publisher = {The Open Journal},
volume = {6},
number = {66},
pages = {3173},
author = {Tiago Tresoldi},
title = {Ngesh: a Python library for synthetic phylogenetic data},
journal = {Journal of Open Source Software}
}
项目详情
下载文件
下载适合您平台的文件。如果您不确定要选择哪个,请了解更多关于安装包的信息。