在napari中使用OpenCL加速图像处理
项目描述
napari-pyclesperanto-assistant
py-clEsperanto-assistant 是一个实验性的 napari 插件,用于构建GPU加速的图像处理工作流程。它是 clEsperanto 项目的一部分,因此,旨在消除生物科学图像处理生态系统中编程语言相关的障碍。它使用 pyclesperanto 和 pyopencl 作为处理图像的后端。
此napari插件向工具菜单中添加了一些菜单项。您可以通过括号中的后缀 (clEsperanto) 来识别它们。此外,它还可以从 napari-assistant 图形用户界面中使用。因此,只需点击菜单 工具 > 通用 > 助手 (na) 或从命令行运行 naparia。
用法
启动助手
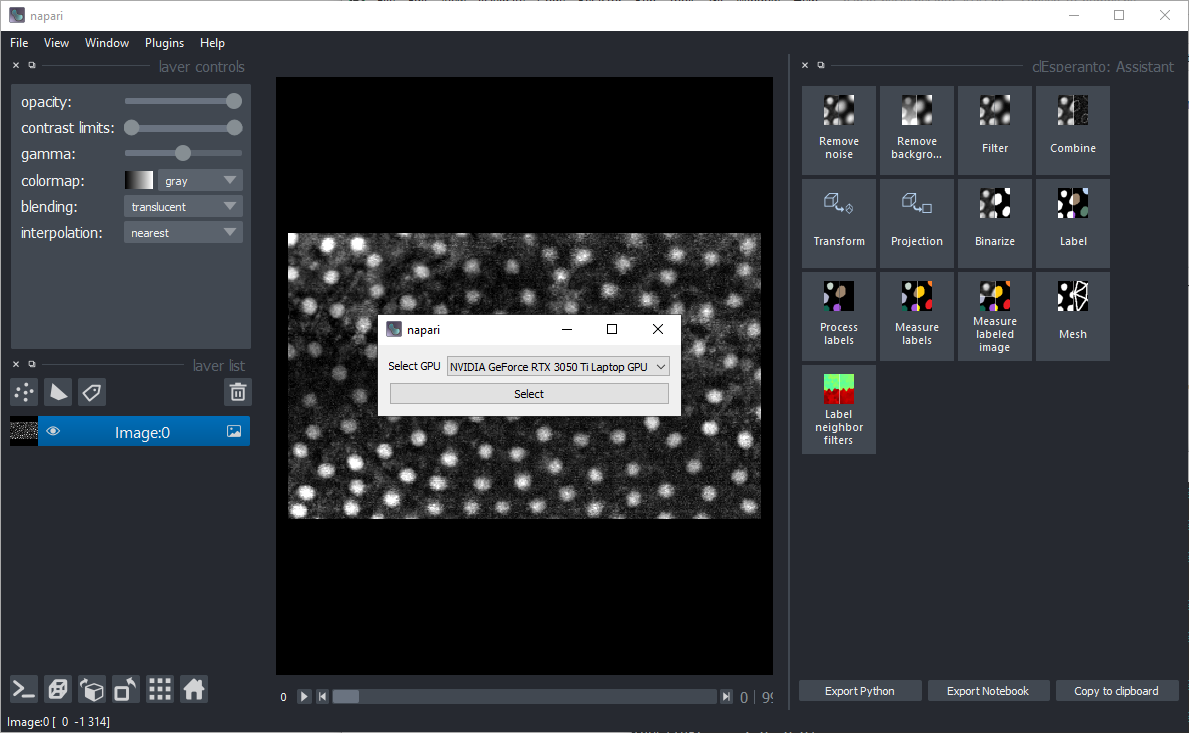
启动napari,例如从命令行
napari
加载示例数据,例如从菜单 文件 > 打开示例 > clEsperanto > CalibZAPWfixed 并从菜单 工具 > 通用 > 助手 (na) 启动助手。
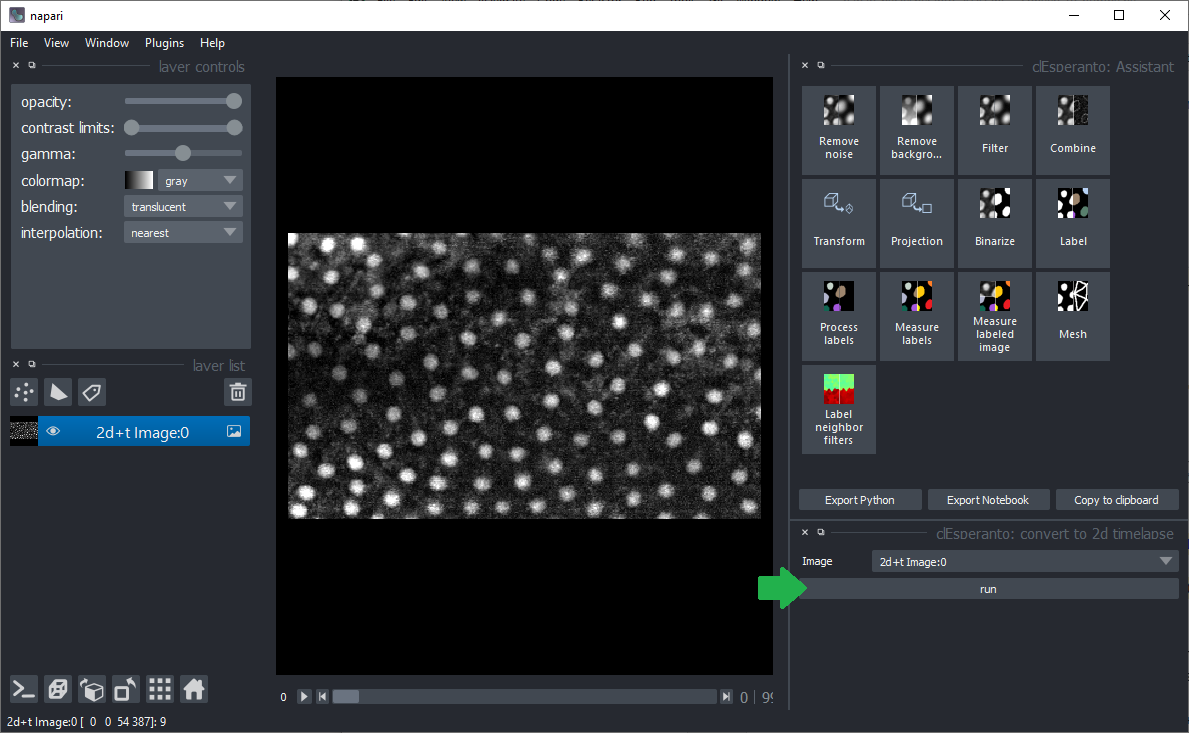
对于二维时间序列数据,根据数据源可能需要进行初步转换步骤。点击菜单 工具 > 工具 > 转换为2D时间序列。在对话框中,选择数据集并点击确定。之后您可以删除原始数据集。
设置工作流程
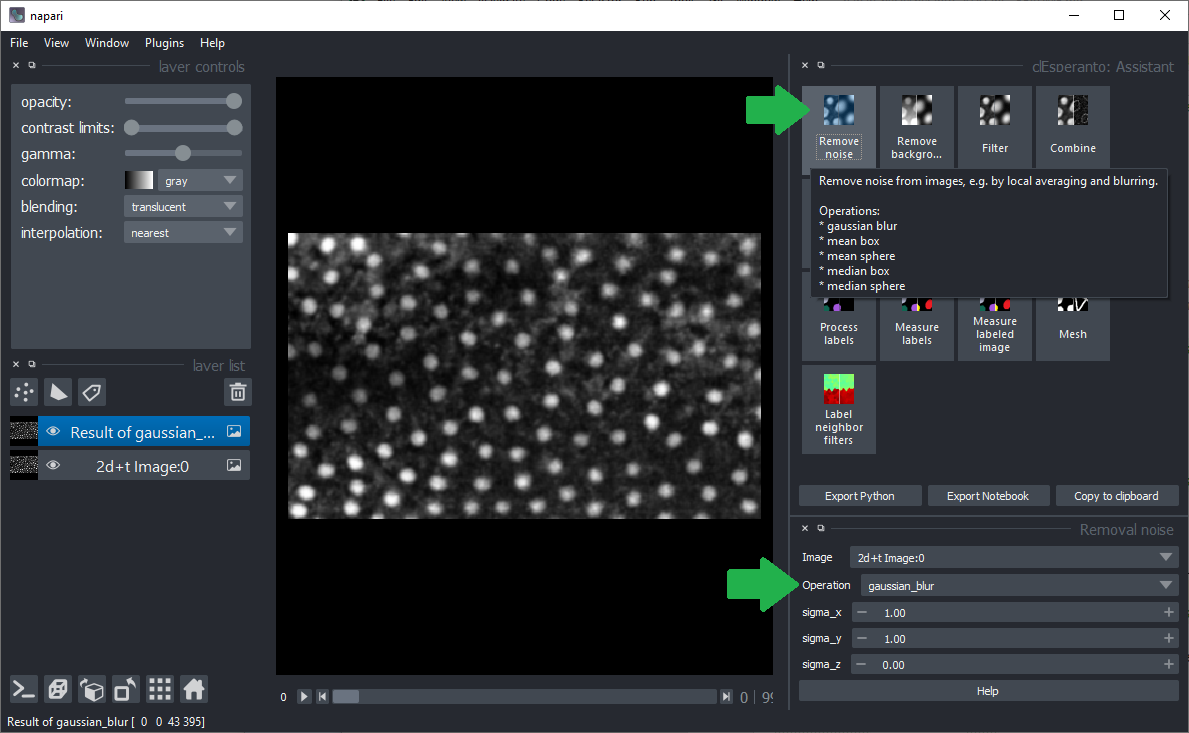
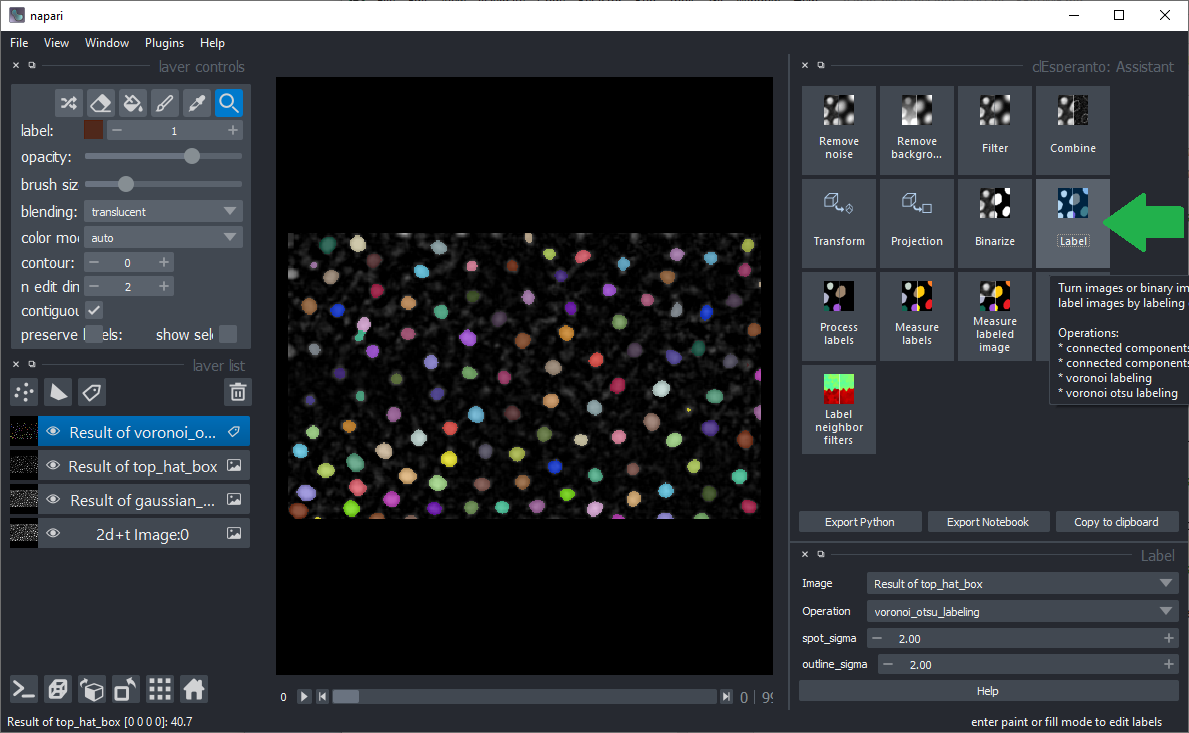
在右上角的面板中选择操作类别,例如,从使用sigma 1的Gaussian模糊进行去噪开始。
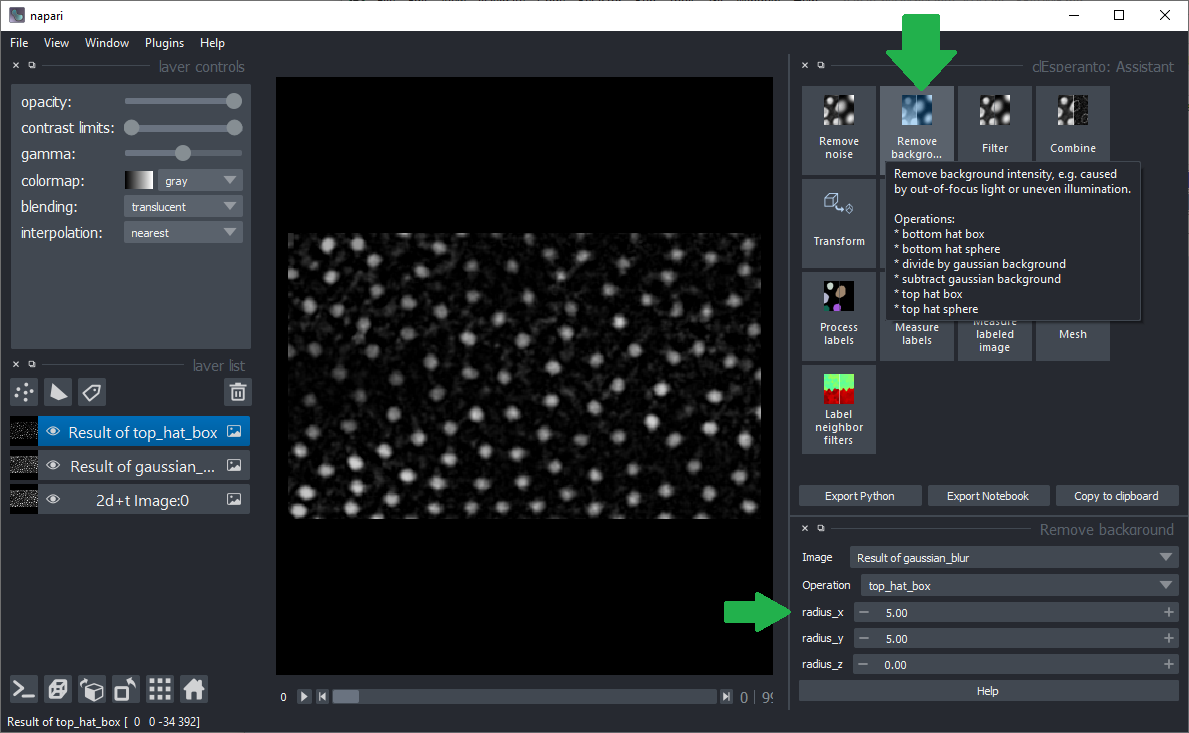
接着使用半径为5的top-hat滤波器去除背景。
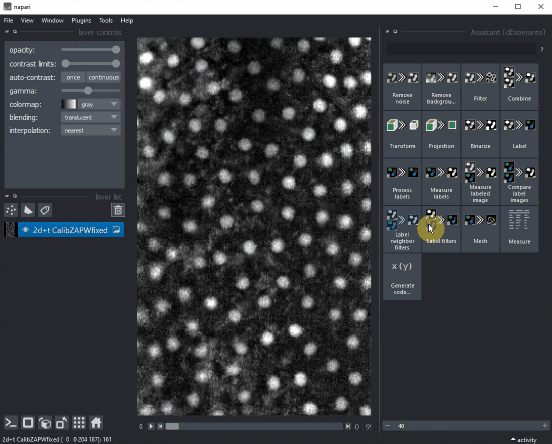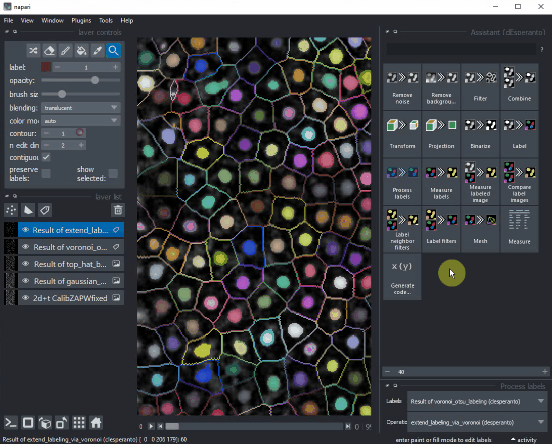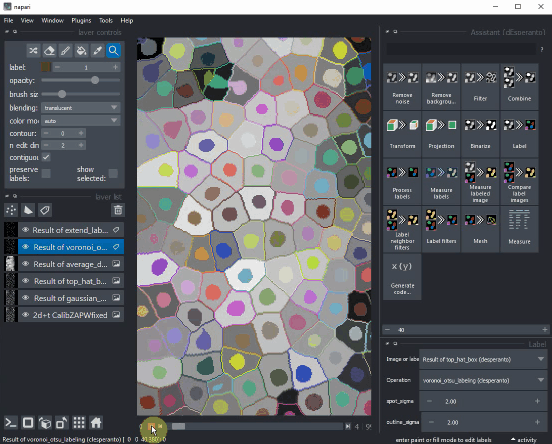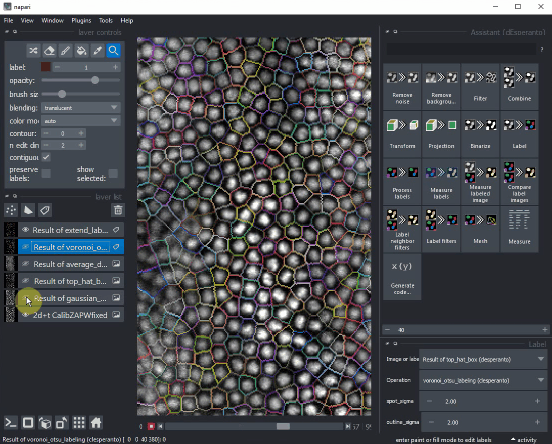
对于对象标注,使用Voronoi-Otsu-Labeling,并将两个sigma参数都设置为2。
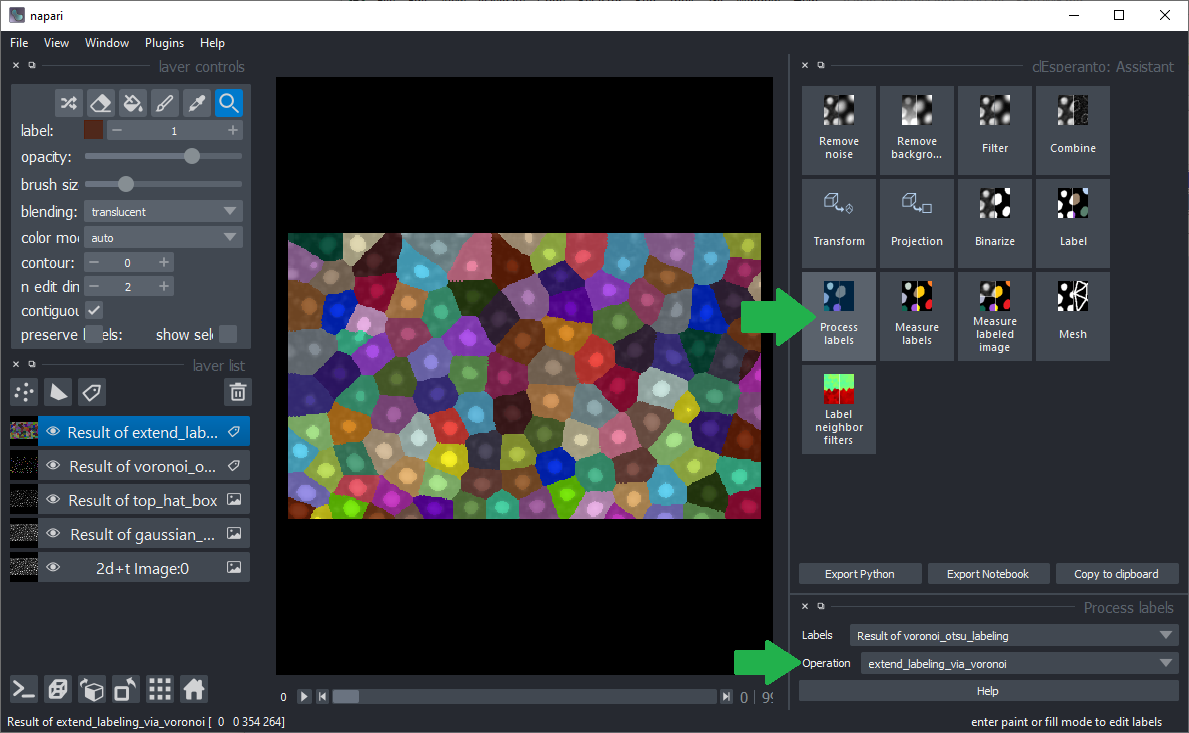
可以使用Voronoi图扩展标注对象,从而得到细胞边界的估计。
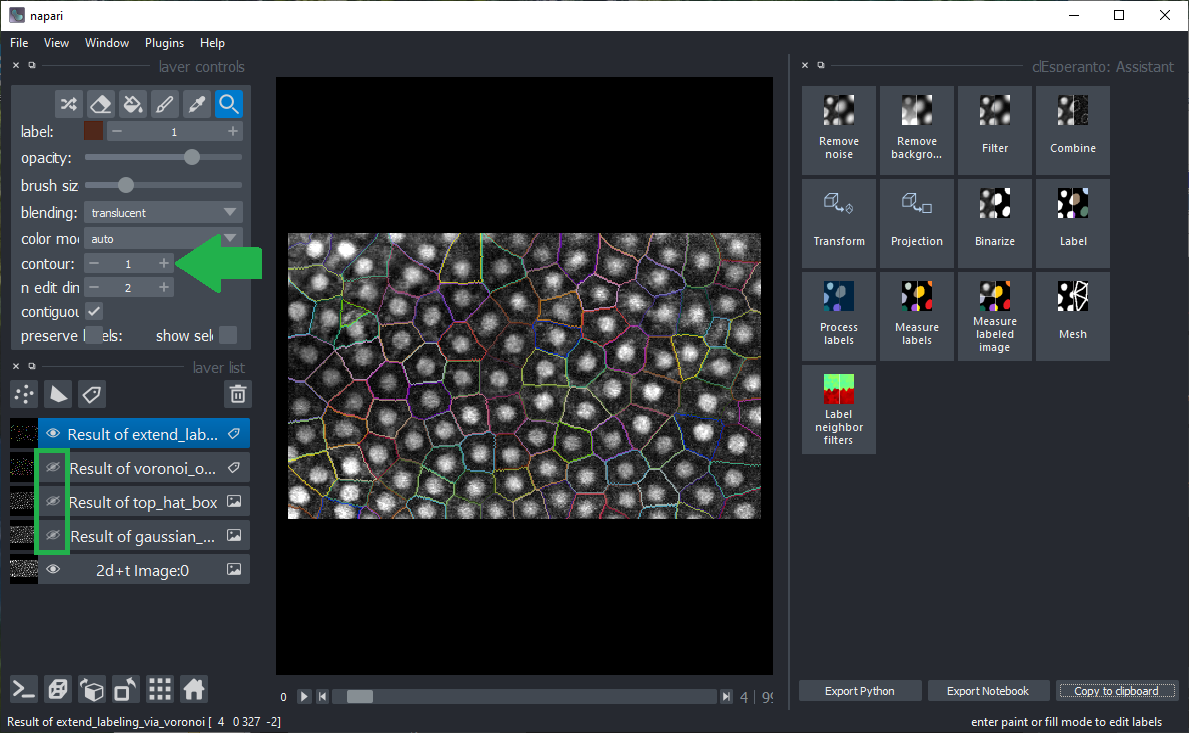
然后您可以配置napari,在原始图像上方显示标签边界。
当您的流程设置好后,点击数据集下方播放按钮。
邻域统计
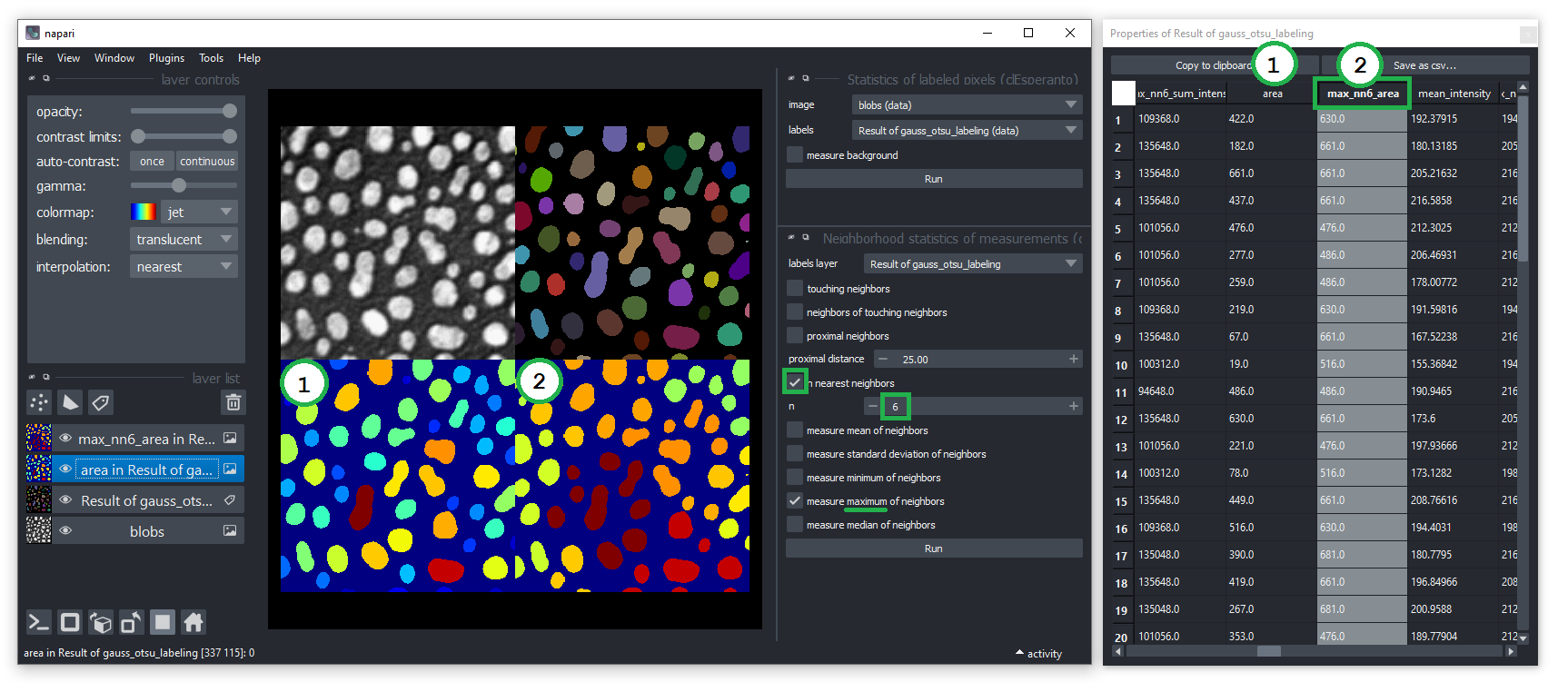
当处理2D或3D数据时,您可以分析测量值与其邻居的关系。例如,您可以使用菜单 工具 > 测量 > 标注像素的统计信息(clesperant) 测量下面示例中显示的blob区域,并通过双击表列(1)将其可视化作为 area 图像。此外,您还可以使用菜单 工具 > 测量 > 测量的邻域统计 测量6个最近邻居的最大面积。新的列将被命名为 "max_nn6_area..."(2)。在相邻的参数图像之间可视化时,建议使用 napari-brightness-contrast 并可视化相同的强度范围,以正确地看到差异。
代码生成
您还可以将您的流程导出为Python/Jython代码或笔记本来使用。有关详细信息,请参阅napari-assistant 文档。
功能
pyclesperanto 提供了各种图像处理可能性。它由从事生命科学研究的开发者开发,因此,它可能专注于处理显示细胞和组织的二维和三维显微镜图像数据。pyclesperanto的部分功能可通过助手用户界面访问。使用此助手可以构建的工作流程示例包括
- 图像滤波
- 去噪/降噪(均值、中值、高斯模糊)
- 背景减除(用于不均匀照明或失焦光)(底部帽子、顶部帽子、减去高斯背景)
- 灰度值形态学(局部最小值、最大值、方差)
- 伽玛校正
- Laplace算子
- Sobel算子
- 组合图像
- 掩码
- 图像数学(图像的加、减、乘、除)
- 绝对/平方差
- 图像变换
- 平移
- 旋转
- 缩放
- 减少堆栈
- 子堆栈
- 图像投影
- 最小/平均/最大/总和/标准差投影
- 图像分割
- 二值化(阈值、局部最大值检测)
- 标注
- 区域化
- 实例分割
- 语义分割
- 检测标签边缘
- 标记点
- 连通分量标注
- Voronoi-Otsu标注
- 二值图像的后处理
- 膨胀
- 腐蚀
- 二值开运算
- 二值闭运算
- 二值与/或/异或
- 标签图像的后处理
- 标签膨胀(扩展)
- 通过Voronoi扩展标签
- 排除边缘上的标签
- 排除大小/值范围内的标签
- 合并接触的标签
- 参数图
- 邻近/接触邻居计数
- 到接触/邻近/n个最近邻居的距离测量
- 像素计数图
- 平均/最大/扩展比图
- 标签测量/参数图的后处理
- 最小/平均/最大/标准差强度图
- 接触/最近的n个邻居/邻居的标准差
- 邻居网格
- 接触邻居
- 最近的n个邻居
- 邻近邻居
- 距离网格
- 基于标签图像的测量
- 2D/3D边界框
- 最小/平均/最大/总和/标准差强度
- 质心
- 质心
- 质心到平均/最大距离(以及扩展比形状描述符)
- 质心到质心到平均/最大距离(以及扩展比形状描述符)
- 邻居的统计(参见相关出版物)
- 代码导出
- Python/Fiji兼容的Jython
- Python Jupyter笔记本
- pyclesperanto脚本
- 细胞分割
- 细胞计数
- 细胞分化
- 组织分类
安装
建议使用conda安装助手。如果您以前从未使用过conda,建议先阅读这篇博客文章。
conda create --name cle_39 python=3.9 napari-pyclesperanto-assistant
conda activate cle_39
Mac用户请也安装此
conda install -c conda-forge ocl_icd_wrapper_apple
Linux用户请也安装此
conda install -c conda-forge ocl-icd-system
然后您可以使用此命令启动napari-assistant
naparia
欢迎反馈和贡献!
clEsperanto开源开发,因为我们相信开源社区。请参阅我们的社区指南。请随意通过github问题或通过image.sc提供反馈。
致谢
该项目得到了德国研究共同体的支持,德国卓越战略——EXC2068 - 德累斯顿工业大学“生命物理学”卓越集群。该项目部分由Chan Zuckerberg Initiative DAF,硅谷社区基金会的一个咨询基金,资助编号2021-240341(Napari插件加速器资助)得以实现。
项目详情
napari_pyclesperanto_assistant-0.22.1.tar.gz的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 6df5e59e105d91f49826d57b2c61091271f62bba9d061dcb9c0c12d3ca000843 |
|
| MD5 | b626d484e90aaae4e7180907ce986a2d |
|
| BLAKE2b-256 | d2f47cb910f032746576a32a4c601575dede769af02eebbb25f67f4cf2d5d7ec |
napari_pyclesperanto_assistant-0.22.1-py3-none-any.whl的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 2bbb9d73c0a526c2c929728ecf449790eb5f69bcb30b5f4416b37e1e640eb80f |
|
| MD5 | 688fc5cae7902d42beaff12f404c8283 |
|
| BLAKE2b-256 | a8c0d93d754baaf6f10b8c455e66db45dbedcb67184cc60e461c1c76f57e4bf0 |