从CZII CryoET数据门户列出、预览和打开数据
项目描述
napari-cryoet-data-portal
在 napari 中列出和打开来自CZ Imaging Institute的 CryoET数据门户 的断层扫描。
安装
您可以使用 pip 安装最新稳定版本
pip install napari-cryoet-data-portal
您还需要单独安装napari作为同一环境中的Python包。包含Qt的一种方法是运行
pip install "napari[all]"
但更普遍的做法是遵循 最新的napari安装说明。
用法
请参阅以下视频,了解插件的基本用法演示。
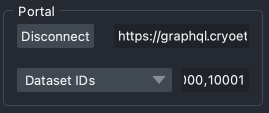
点击 连接 按钮以建立与数据门户的连接。
您可以选择查询数据集、运行、体素间距或断层扫描的子集,使用它们的相应ID。这可以在门户增长时加快列表过程。要这样做,请从此面板关联的下拉框中选择ID类型,然后在旁边的文本框中输入感兴趣的ID,用逗号分隔。例如,如果您只想列出数据集10000和10001,请从下拉框中选择数据集ID,然后在文本框中输入文本10000,10001。默认情况下,列出所有数据集。
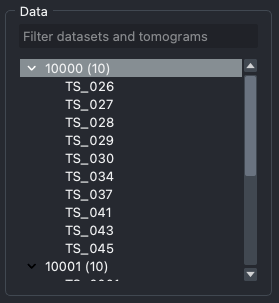
连接到门户后,找到的数据集将如下添加。
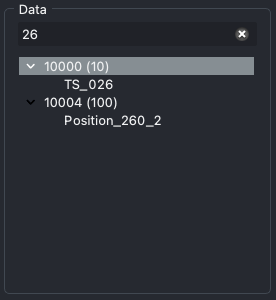
可以通过指定正则表达式模式来过滤数据集和断层扫描。
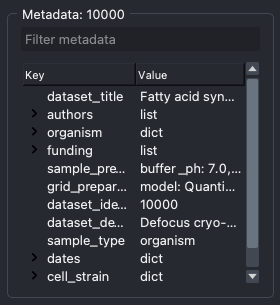
选择数据集将显示其元数据,可以对其进行类似探索和过滤。
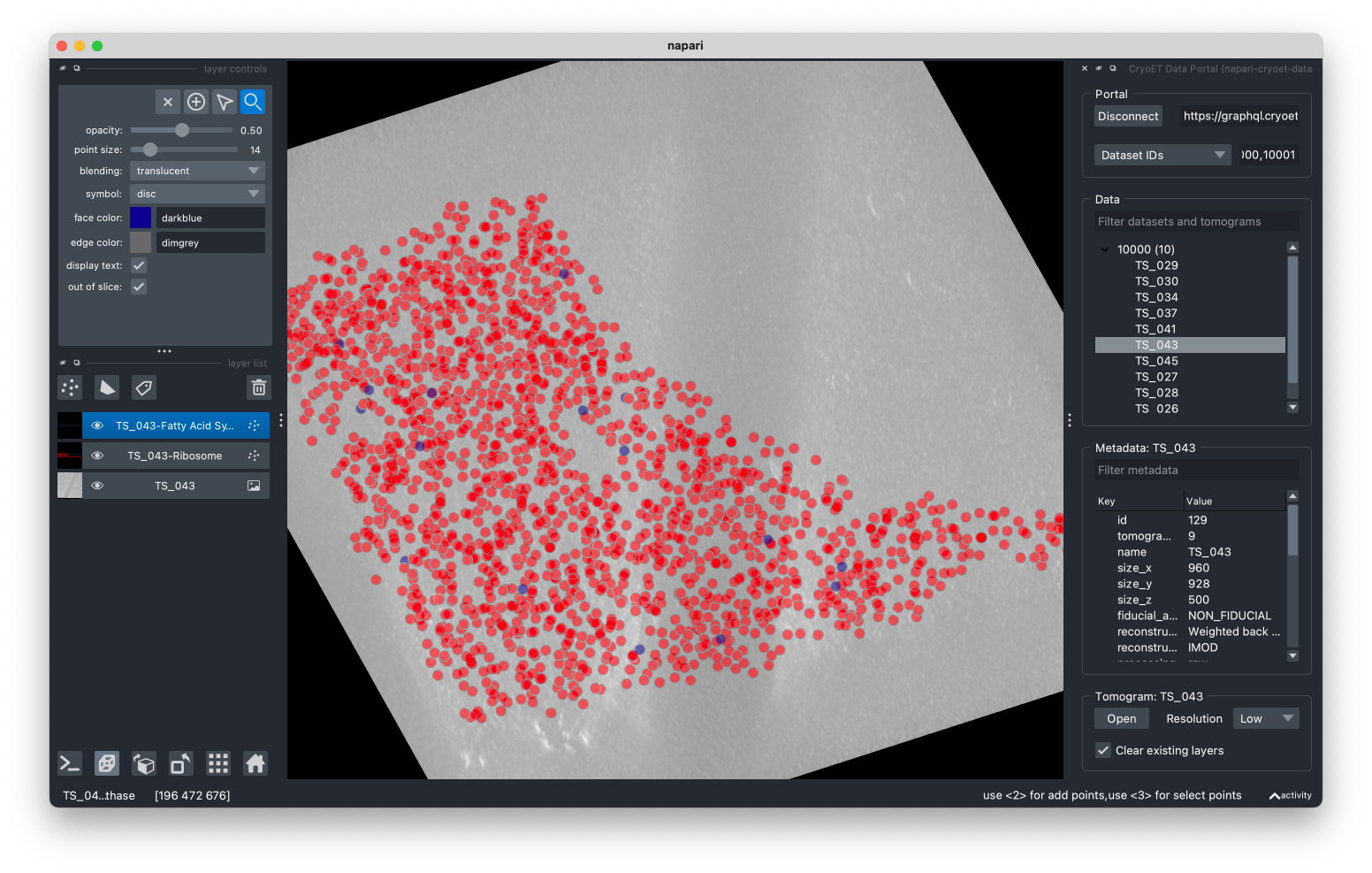
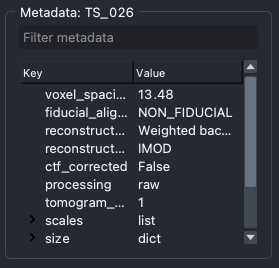
选择断层扫描将显示其元数据,并在napari查看器中打开最低分辨率的断层扫描及其所有相关点注释。
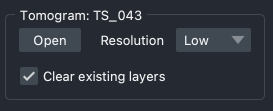
通过选择不同的分辨率并单击打开按钮,可以加载更高分辨率的断层扫描。
在这种情况下,napari仅加载画布上需要显示的数据。虽然这可以减少加载的数据量,但在初次打开和探索数据时可能会引起性能问题。默认情况下,打开新的断层扫描会清除napari中所有现有图层。如果您想要保留这些图层,请取消选中此面板中的相关复选框。
一般来说,从门户查找和获取数据可能需要很长时间。所有从门户获取数据的插件操作都尝试并行运行,以尽可能使与napari和插件的交互保持响应。这些操作也可以通过单击取消按钮来取消。
贡献
这仍在早期开发中,但欢迎贡献和想法!请不要犹豫,打开一个问题或打开一个拉取请求以帮助改进此插件。
要设置开发环境,请Fork此存储库,克隆您的分支,切换到其顶层目录并运行
pip install -e ".[testing]"
本项目遵循贡献者公约行为准则。通过参与,您应遵守此准则。请向opensource@chanzuckerberg.com报告不可接受的行为。
安全
如果您认为您发现了一个安全问题,请参阅我们的安全策略,了解如何报告。
许可证
根据MIT许可证分发,"napari-cryoet-data-portal"是免费和开源软件。有关更多详细信息,请参阅许可证文件。
致谢
此插件是用Cookiecutter和@napari的cookiecutter-napari-plugin模板生成的。
项目详情
下载文件
下载适用于您平台的应用程序。如果您不确定选择哪个,请了解有关安装软件包的更多信息。
源代码分发
构建分发
napari_cryoet_data_portal-0.4.1.tar.gz 的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | c9b30c9f22ebfd8b237e3a65f56479ca1894415393d39b20e0560c62618027ea |
|
| MD5 | a1e9e22e33a045d85757bcf684d79f32 |
|
| BLAKE2b-256 | 080fb97bf585a271a8aea26416e220c5948264891baad28bd0b84919bf22ab9a |
napari_cryoet_data_portal-0.4.1-py3-none-any.whl 的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 09ab9ca80b4e7cb1204aa1be2578de489ab51ebec23aa20814269a7c39d39556 |
|
| MD5 | 11a5c5ea577f16ccfb500f6967eb18be |
|
| BLAKE2b-256 | 4fe440beb43f065e6b5f8d532498e550976a205ab301f27f176b908ca710a28d |