基于BrainWeb的20个正常脑的多模态模型
项目描述
以下示例可以通过以下方式交互式启动:
基于BrainWeb的20个正常脑的多模态模型
本项目最初受到“BrainWeb:20个正常脑的解剖模型”的启发。
然而,本项目还包括许多通用的工具、图像处理和显示功能。例如,包括用于交互式比较多个3D体积的 volshow(),用于缓存数据URL的 get_file(),以及用于图像配准的 register()。
PET-MR模拟的下载和预处理
如果文件已存在,此笔记本将不会重新下载/重新处理文件。
输出数据
~/.brainweb/subject_*.npz:数据类型(shape):float32(127, 344, 344)
-
~/.brainweb/subject_*.bin.gz:数据类型(shape):uint16(362, 434, 362)
安装
pip安装brainweb
作者:Casper da Costa-Luis <casper.dcl@physics.org>
日期:2017-2020
许可证: MPLv2.0
from __future__ import print_function, division
%matplotlib notebook
import brainweb
from brainweb import volshow
import numpy as np
from os import path
from tqdm.auto import tqdm
import logging
logging.basicConfig(level=logging.INFO)原始数据
# download
files = brainweb.get_files()
# read last file
data = brainweb.load_file(files[-1])
# show last subject
print(files[-1])
volshow(data, cmaps=['gist_ncar']);~/.brainweb/subject_54.bin.gz
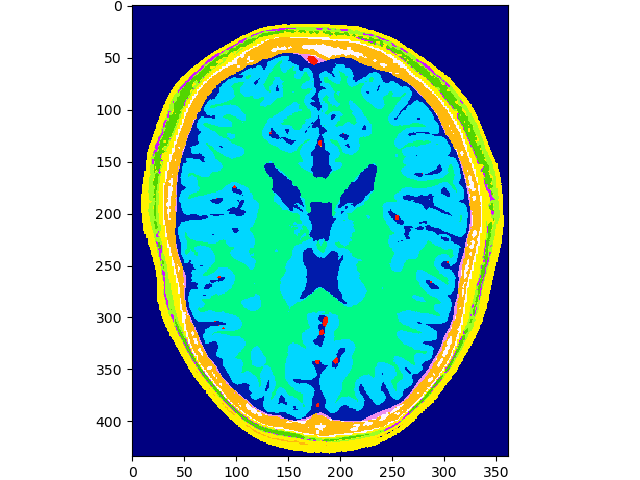
转换
转换原始图像数据
西门子Biograph mMR分辨率(~2mm)及尺寸(127, 344, 344)
PET/T1/T2/uMap强度
PET默认为FDG强度比;可以使用例如淀粉样蛋白替代
PET/T1/T2的随机结构
t = (1 + g [2 G_sigma(r) - 1]),其中
r = rand(127, 344, 344)在[0, 1)区间内,
高斯平滑sigma = 1,
g = 1用于PET;0.75用于MRI,并且
t = PET或MRI的分段常数幻影
# show region probability masks
PetClass = brainweb.FDG
label_probs = brainweb.get_label_probabilities(files[-1], labels=PetClass.all_labels)
volshow(label_probs[brainweb.trim_zeros_ROI(label_probs)], titles=PetClass.all_labels, frameon=False);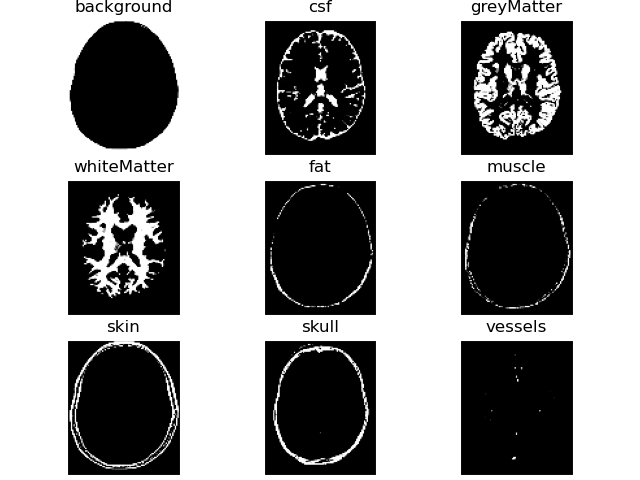
brainweb.seed(1337)
for f in tqdm(files, desc="mMR ground truths", unit="subject"):
vol = brainweb.get_mmr_fromfile(
f,
petNoise=1, t1Noise=0.75, t2Noise=0.75,
petSigma=1, t1Sigma=1, t2Sigma=1,
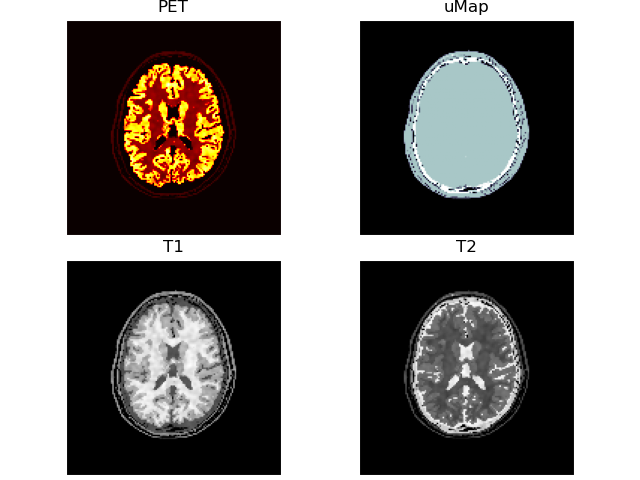
PetClass=PetClass)# show last subject
print(f)
volshow([vol['PET' ][:, 100:-100, 100:-100],
vol['uMap'][:, 100:-100, 100:-100],
vol['T1' ][:, 100:-100, 100:-100],
vol['T2' ][:, 100:-100, 100:-100]],
cmaps=['hot', 'bone', 'Greys_r', 'Greys_r'],
titles=["PET", "uMap", "T1", "T2"],
frameon=False);~/.brainweb/subject_54.bin.gz
# add some lesions
brainweb.seed(1337)
im3d = brainweb.add_lesions(vol['PET'])
volshow(im3d[:, 100:-100, 100:-100], cmaps=['hot']);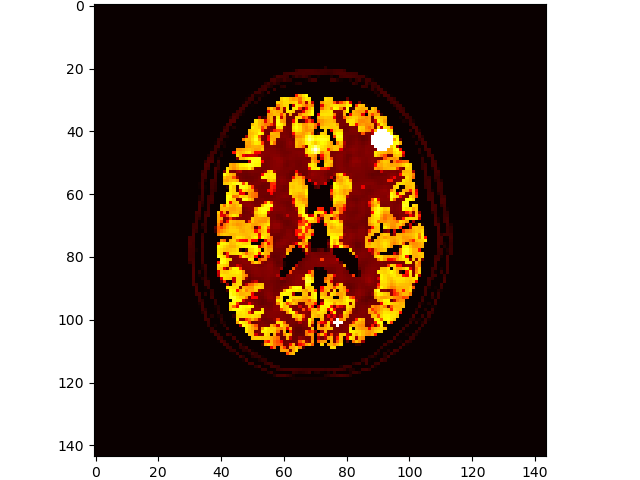
# bonus: use brute-force registration to transform
#!pip install -U 'brainweb[register]'
reg = brainweb.register(
data[:, ::-1], target=vol['PET'],
src_resolution=brainweb.Res.brainweb,
target_resolution=brainweb.Res.mMR)
volshow({
"PET": vol['PET'][:, 100:-100, 100:-100],
"RawReg": reg[ :, 100:-100, 100:-100],
"T1": vol['T1' ][:, 100:-100, 100:-100],
}, cmaps=['hot', 'gist_ncar', 'Greys_r'], ncols=3, tight_layout=5, figsize=(9.5, 3.5), frameon=False);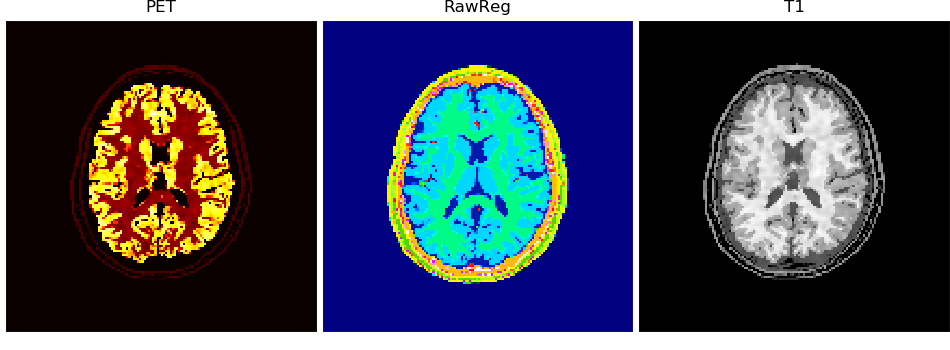
项目详情
下载文件
下载适用于您平台的文件。如果您不确定该选择哪个,请了解有关安装包的更多信息。
源分发
brainweb-1.6.2.tar.gz (13.6 kB 查看散列值)
构建分发
brainweb-1.6.2-py2.py3-none-any.whl (11.5 kB 查看散列值)
关闭
brainweb-1.6.2.tar.gz的散列值
| 算法 | 散列摘要 | |
|---|---|---|
| SHA256 | cfca5a08d519326662d4135034f340cee7a664e1e02d85bb348c217baeb74eaf |
|
| MD5 | 44f8763fb75f9b114b9bf8789cb012c0 |
|
| BLAKE2b-256 | ee4cc26866bc11ee2d82c3592ee9c67afe6d67e9a566b1490cf1f103a4489e13 |
关闭
brainweb-1.6.2-py2.py3-none-any.whl的散列值
| 算法 | 散列摘要 | |
|---|---|---|
| SHA256 | db7d16ccc7d5f1dd02094a5eeb991b1554a47fceac34bfab3d6cfb634a7752f1 |
|
| MD5 | 2aad724a3b75c666f7d83b751e4795ef |
|
| BLAKE2b-256 | d882e9a43fb3468158dfe6d7a1b6e642bc243abc16fc02175f7491abae990394 |









