Pydra数据流引擎
项目描述
pydra-ml
Pydra-ML是一个利用Pydra和scikit-learn进行一组分类器之间模型比较的演示应用程序。目的是使用此应用程序使Pydra更健壮,同时允许用户更轻松地生成分类报告。此应用程序利用Pydra强大的拆分器和组合器来扩展到一组分类器和指标。它还将使用Pydra的缓存来
-
使用嵌套重采样(在内循环中执行k折交叉验证以进行超参数调整)高效地训练模型
-
比较一些scikit-learn流水线以及基本分类器(即,显示不同模型性能的分布)。
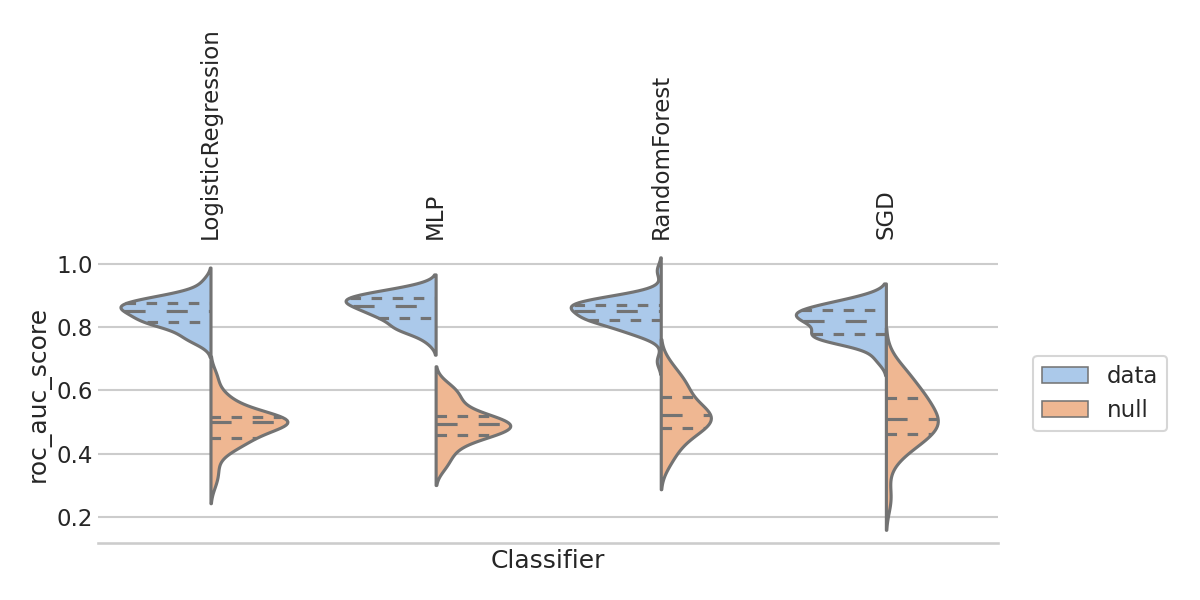

-
当添加新指标或增加迭代次数(
n_splits)时,保存模型而无需重新进行模型训练和评估。只需更改规范文件,它将使用存储的模型来节省时间。 -
输出报告包含三种类型的 特征重要性 方法
-
(1) sklearn中一些模型的标准特征重要性方法(例如,线性模型的
coef_,基于树的模型的feature_importances_),未完全测试 -
(2) sklearn的 排列重要性(模型无关,适用于所有模型),未完全测试
-
(3) Kernel SHAP特征重要性(模型无关,适用于所有模型)
数据集的每个自助法划分可能会创建其自己的模型(例如,不同的权重或最佳超参数)。对于每个划分,我们取所有测试预测的绝对SHAP值的平均值。然后,我们计算所有划分的SHAP值的平均值。
安装
pydraml需要Python 3.7以上版本。
pip install pydra-ml
命令行界面使用
此存储库安装了pydraml命令行界面,以便无需编程即可使用。
要测试分类示例的CLI,将pydra_ml/tests/data/breast_cancer.csv和examples/classification_cancer_short-spec.json复制到文件夹中,并在examples/文件夹中运行或在此文件夹中运行。
$ pydraml -s classification_cancer_short-spec.json
目前,gen_feature_importance和gen_permutation_importance仅与线性模型一起工作。我们需要在其他模型上测试,包括具有超参数调整的管道。
$ pydraml -s classification_cancer_toy-spec.json
要检查回归示例,将pydra_ml/tests/data/diabetes_table.csv和examples/regression_diabetes_spec.json复制到文件夹中,并在examples/文件夹中运行或在此文件夹中运行。
$ pydraml -s regression_diabetes_spec.json
对于每个案例,pydra-ml将生成一个结果文件夹out-{spec_file_name}-{timestamp}/,其中包含比较每个模型及其重要特征的图形和表格,以及包含模型评估所有得分的pickle结果文件(请参阅下面的输出部分)。
$ pydraml --help
Usage: pydraml [OPTIONS]
Options:
-s, --specfile PATH Specification file to use [required]
-p, --plugin TEXT... Pydra plugin to use [default: cf, n_procs=1]
-c, --cache TEXT Cache dir [default:
/Users/satra/software/sensein/pydra-ml/cache-wf]
--help Show this message and exit.
使用插件选项,您可以使用本地多进程
$ pydraml -s ../classification_cancer_short-spec.json -p cf "n_procs=8"
或通过dask执行。
$ pydraml -s ../classification_cancer_short-spec.json -p dask "address=tcp://192.168.1.154:8786"
当前规范
当前规范是以下示例中显示的JSON文件。它需要包含此处描述的所有字段。对于具有许多特征的数据库,您需要程序化生成x_indices。
- filename:包含数据的CSV文件的绝对路径。可以包含一个名为
group的列,以支持GroupShuffleSplit,否则每个样本被视为一个组。 - x_indices:用作输入特征的列名列表(数字或字符串)。请注意,不要在此处包含输出或目标变量。
- target_vars:目标变量的字符串列表(目前仅支持一个)
- group_var:表示分组列的字符串
- n_splits:要使用的洗牌划分迭代次数
- test_size:每个迭代中用于测试集的数据比例
- clf_info:要使用的scikit-learn分类器的列表
- permute:布尔值列表,指示是否生成带有排列标签的空模型(即排列测试)(设置为true)或不是(设置为false)
- gen_feature_importance:布尔值,指示是否应生成每个模型独有的特征重要性方法(如果可用)(例如,线性模型的
coef_,基于树的模型的feature_importances_)未完全测试:设置为false - gen_permutation_importance: 布尔值,表示是否生成排列重要性值(模型无关,适用于所有模型)未完全测试:设置为false
- gen_shap: 布尔值,表示是否生成SHAP值(模型无关,适用于所有模型)
- nsamples: 用于SHAP估计的样本数量,使用整数或“auto”设置则使用
nsamples = 2 * X.shape[1] + 2048。 - l1_reg: 用于SHAP估计的正则化器类型
- plot_top_n_shap: 要绘制的最高SHAP值的数量或比例(例如,16或0.1表示最高10%)。设置为1.0(浮点数)以绘制所有特征或1(整数)以绘制最高第一个特征。
- metrics: 要使用的scikit-learn度量
clf_info规范
这是一个来自scikit learn的分类器列表,并使用数组进行编码
- module
- classifier
- (optional) classifier parameters
- (optional) gridsearch param grid
当提供参数网格且默认分类器参数未更改时,则必须提供空字典作为参数3。
这也可以嵌入为列表,表示scikit-learn的Pipeline。例如
[ ["sklearn.impute", "SimpleImputer"],
["sklearn.preprocessing", "StandardScaler"],
["sklearn.tree", "DecisionTreeClassifier", {"max_depth": 5}]
]
示例规范
{"filename": "breast_cancer.csv",
"x_indices": ["radius_mean", "texture_mean","perimeter_mean", "area_mean", "smoothness_mean",
"compactness_mean", "concavity_mean", "concave points_mean",
"symmetry_mean", "fractal_dimension_mean", "radius_se",
"texture_se", "perimeter_se", "area_se", "smoothness_se",
"compactness_se", "concavity_se", "concave points_se",
"symmetry_se", "fractal_dimension_se", "radius_worst",
"texture_worst", "perimeter_worst", "area_worst",
"smoothness_worst", "compactness_worst", "concavity_worst",
"concave points_worst", "symmetry_worst", "fractal_dimension_worst"],
"target_vars": ["target"],
"group_var": null,
"n_splits": 100,
"test_size": 0.2,
"clf_info": [
["sklearn.ensemble", "AdaBoostClassifier"],
["sklearn.naive_bayes", "GaussianNB"],
[ ["sklearn.impute", "SimpleImputer"],
["sklearn.preprocessing", "StandardScaler"],
["sklearn.tree", "DecisionTreeClassifier", {"max_depth": 5}]],
["sklearn.ensemble", "RandomForestClassifier", {"n_estimators": 100}],
["sklearn.ensemble", "ExtraTreesClassifier", {"n_estimators": 100, "class_weight": "balanced"}],
["sklearn.linear_model", "LogisticRegressionCV", {"solver": "liblinear", "penalty": "l1"}],
["sklearn.neural_network", "MLPClassifier", {"alpha": 1, "max_iter": 1000}],
["sklearn.svm", "SVC", {"probability": true},
[{"kernel": ["rbf", "linear"], "C": [1, 10, 100, 1000]}]],
["sklearn.neighbors", "KNeighborsClassifier", {},
[{"n_neighbors": [3, 5, 7, 9, 11, 13, 15, 17, 19],
"weights": ["uniform", "distance"]}]]
],
"permute": [true, false],
"gen_feature_importance": false,
"gen_permutation_importance": false,
"permutation_importance_n_repeats": 5,
"permutation_importance_scoring": "accuracy",
"gen_shap": true,
"nsamples": "auto",
"l1_reg": "aic",
"plot_top_n_shap": 16,
"metrics": ["roc_auc_score", "f1_score", "precision_score", "recall_score"]
}
输出
工作流程将输出:<<<<<<< HEAD
-
results-{timestamp}.pkl包含每个使用的模型的1个列表。例如,如果pkl文件分配给变量results,则通过results[0]到results[N]访问模型。如果permute: [false,true],则将首先输出在标签上训练的模型(results[0])和其次在排列标签上训练的模型(results[1])。如果有额外模型,则将通过results[2](标签)和results[3](排列)访问。每个模型包含
-
通过
results[0][0]访问的dict,其中包含模型信息import pickle as pk with open("results-20201208T010313.229190.pkl", "rb") as fp: results = pk.load(fp) print(results[0][0]) #1st model trained on labels
{'ml_wf.clf_info': ['sklearn.neural_network', 'MLPClassifier', {'alpha': 1, 'max_iter': 1000}], 'ml_wf.permute': False}print(results[3][0]) #2nd models trained on permuted labels
{'ml_wf.clf_info':['sklearn.linear_model', 'LogisticRegression', {'penalty': 'l2'}], 'ml_wf.permute': True} -
通过
results[0][1].output访问的pydra Result obj:=======
-
-
results-{timestamp}.pkl包含每个使用的模型的1个列表。例如,如果分配给变量results,则通过results[0]到results[N]访问。如果permute: [true,false],则将首先输出在排列标签上训练的模型(results[0])和其次在标签上训练的模型(results[1])。如果有额外模型,则将通过results[2]和results[3]访问。每个模型包含- 通过
results[0][0]访问的dict,其中包含模型信息:{'ml_wf.clf_info': ['sklearn.neural_network', 'MLPClassifier', {'alpha': 1, 'max_iter': 1000}], 'ml_wf.permute': False} - 通过
results[0][1]访问的具有属性output的pydra Result obj
- 通过
ea2092bb5f199aa6ff83f25f863d3652f824f6af,它本身具有属性:-
feature_names:来自数据csv的列。
```python
print(results[1][1].output.feature_names)
```
`['mean radius', 'mean texture', 'mean perimeter', 'mean area', ... ]`
And the following attributes organized in *n_splits* lists for *n_splits* bootstrapping samples:
- `output`: *n_splits* lists, each one with two lists for true and predicted labels.
- `score`: *n_splits* lists each one containing M different metric scores.
Three types of feature importance methods:
- (1) `feature_importance`: standard feature importance method from *sklearn*. Limitation: not all models have standard methods and difficult to compare methods across models.
- `pipeline.coef_` for linear models (coefficients of regression, SVC).
- `pipeline.coefs_` for multi-layer perceptron, which returns `j` lists for `j` hidden nodes connections with each input
- `pipeline.feature_importances_` for decision tree, Random Forest, or boosting algorithms
```python
print(results[1][1].output.feature_importance)
```
- (2) `permutation_importance`: the difference in performance from permutating the feature column as in [sklearn's permutation importance](https://scikit-learn.cn/stable/modules/generated/sklearn.inspection.permutation_importance.html).
Advantage: works for all models (i.e., model agnostic). Limitation: measures decrease in performance, not magnitude of each feature.
```python
print(results[1][1].output.permutation_importance)
```
- (3) `shaps`: `n_splits` lists each one with a list of shape (P,F) where P is the
amount of predictions and F the different SHAP values for each feature.
`shaps` is empty if `gen_shap` is set to `false` or if `permute` is set
to true. Advantage: model agnostic, produces magnitude for each feature.
```python
print(results[1][1].output.shaps)
```
- `model`: A pickled version of the model trained on all the input data.
One can use this model to test on new data that has the exact same input
shape and features as the trained model. For example:
```python
import pickle as pk
import numpy as np
with open("results-20201208T010313.229190.pkl", "rb") as fp:
results = pk.load(fp)
trained_model = results[0][1].output.model
trained_model.predict(np.random.rand(1, 30))
```
Please make sure the value of `results[N][0].get('ml_wf.permute')` is `False` to ensure that you are not using
a permuted model.
- 每个度量一个图,展示性能在各个拆分(有无null分布训练在排列标签上)中的分布
performance_table-{timestamp}文件夹test-performance-table_{metric}_all-splits_{timestamp).csv包含每个在各个bootstrapping拆分上训练的模型/s的测试性能和平均值test-performance-table_accuracy_score_with-95ci-and-median-null_20210702T223005.935447test-performance-table_{metric}_all-splits_{timestamp).csv包含平均值、95%置信区间(CI)和如果有null模型则包含平均值:平均值 [95% CI; null模型平均值]
stats-{metric}-{timestamp}.png:包含带有关键词score的任何指标的一个图表,使用经验p值进行单尾统计比较(行 > 列),评估分类器性能的常用有效措施(参见Ojala & Garriga,2010年的定义1),在sklearn中实现。注释 = p值,颜色 = 超过0.05的alpha水平显著。p值表示列模型分数中行模型分类器具有更高平均性能的分数比例(例如,p值为0.02表示行模型的平均分数高于98%的列模型分数)。对角线上显示数据模型与空模型。实际数值存储在相应命名的pkl文件中。shap-{timestamp}目录- 为每个分割的测试集中的每个预测计算SHAP值(例如,30个重抽样分割和100个预测将创建一个(30,100)数组)。对每个分割的预测取平均值(例如,对于64个特征和30个重抽样样本,结果将是一个(64,30)数组)。
- 对于二进制分类,通过将预测分割为TP、TN、FP和FN来更准确地显示特征重要性,这反过来又允许进行错误审计(即模型在做出错误/虚假预测时关注的内容)
quadrant_indexes.pkl:将TP、TN、FP、FN索引保存为一个包含每个模型一个键的dict(没有SHAP值的排列模型将自动跳过),每个键的值是一个重抽样分割。summary_values_shap_{model_name}_{prediction_type}.csv包含所有SHAP值和按重抽样分割的均值SHAP值排名的汇总统计信息。如果这个分割没有在文件名中包含该类型的预测(例如,您可能没有FN或FP在性能高的分割中),则sample_n列可能为空或NaN。summary_shap_{model_name}_{plot_top_n_shap}.png包含所有特征(设置为1.0)或仅包含前N个最重要的特征的SHAP值汇总统计信息,以更好地进行可视化。
调试
现在您需要了解一些pydra才能了解如何调试此应用程序。如果进程崩溃,最简单的方法是先删除cache-wf文件夹。然而,如果您正在重新运行,您也可以删除cache-wf目录中的任何.lock文件。
开发者安装
以开发者模式安装repo
git clone https://github.com/nipype/pydra-ml.git
cd pydra-ml
pip install -e .[dev]
安装pre-commit也非常有用,它在提交代码时负责处理样式。当使用pre-commit时,您可能需要运行git commit两次,因为pre-commit可能会对您的代码进行额外的样式更改,并且默认情况下不会提交这些更改。
pip install pre-commit
pre-commit install
项目结构
tasks.py包含Python函数。classifier.py包含Pydra工作流程和注释任务。report.py包含报告生成代码。
项目详情
下载文件
下载适合您平台的自定义文件。如果您不确定选择哪一个,请了解有关安装包的更多信息。
源分布
构建分发
pydra_ml-0.7.0.tar.gz 的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 3c48555336dfb790c9a43f97cd605a96629695f0f72b9c8da7da889e5a83bab5 |
|
| MD5 | d12f552ab33fb3a6a8be2ddf191d2bf3 |
|
| BLAKE2b-256 | d379e4bf7d2888e4f568a5f76702a491af678b510c60cd063c9a980b0517d074 |
pydra_ml-0.7.0-py3-none-any.whl 的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 9227ed2259759540b9d3566c012128c60781482cc92d68f7ced54a46d695f95d |
|
| MD5 | 80222babb00e218360f4bd6069e5ad88 |
|
| BLAKE2b-256 | 2ef6b8b047c74a400aa8e9564dc61d8cb630cb1b338fec6769b70ba16507b24f |





