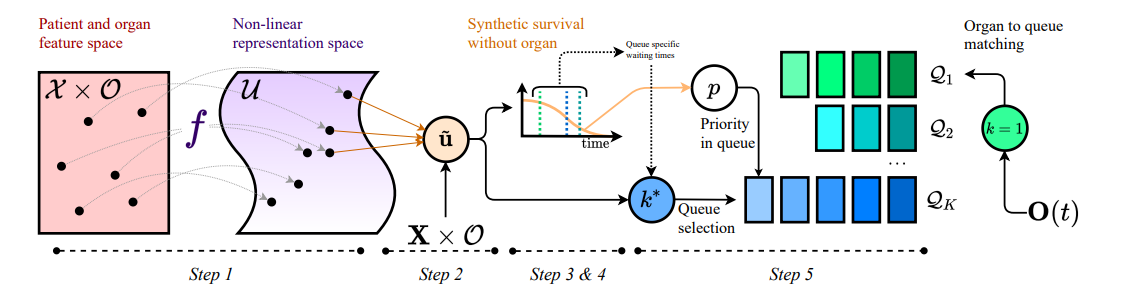
使用可解释反事实生存分析学习器官移植分配的排队策略
项目描述
使用可解释反事实生存分析学习器官移植分配的排队策略
J. Berrevoets, A. M. Alaa, Z. Qian, J. Jordon, A. E. S. Gimson, M. van der Schaar [ICML 2021]
在这个仓库中,我们提供了我们ICML21论文中介绍的OrganSync,一个新颖的器官到患者分配系统的代码。请注意,此代码仅供研究目的使用,不打算用于实际应用。
在我们的论文中,我们对OrganITE进行了基准测试,这是我们之前介绍的一篇论文。我们使用此仓库中相同的框架重新实现了OrganITE(以及其他基准),以便所有代码都是可比较的。对于所有之前的实现,我们参考了OrganITE的专用仓库。
代码作者:J. Berrevoets (jb2384@cam.ac.uk)
仓库结构
此仓库组织如下
organsync/
|- src/
|- organsync/ # Python library core
|- data/ # code to preprocess data
|- eval_policies/ # code to run allocation simulations
|- models/ # code for inference models
|- experiments/
|- data # data modules
|- models # training logic for models
|- notebooks/wandb
|- simulation_tests.ipynb # experiments in Tab.1
|- a_composition # experiments in Fig.3
|- sc_influence.ipynb # experiments in Fig.4, top row
|- rep_influence.ipynb # experiments in Fig.4, bottom row
|- test # unit tests
|- data # datasets
安装
我们提供了一个requirements.txt文件
pip install -r requirements.txt
pip install .
请在新创建的虚拟环境中使用上述文件以避免依赖冲突。所有代码均针对python 3.8.6编写。
可用模型
| 模型 | 论文 | 代码 |
|---|---|---|
| Organsync | 使用可解释反事实生存分析学习器官移植分配的排队策略 | 代码 |
| OrganITE | OrganITE: 使用个体治疗效果进行最优移植器官提供 | 代码 |
| TransplantBenefit | 政策和指导 | 代码 |
| MELD | 预测经肝内门体分流术后患者预后不良的模型 | 代码 |
| MELDna | 肝脏移植等待名单患者低钠血症和死亡率 | 代码 |
| MELD3 | MELD 3.0:适用于现代时代的终末期肝病模型更新 | 代码 |
| UKELD | 英国肝移植患者选择和捐赠器官分配 | 代码 |
使用的框架
我们广泛使用Weights and Biases(W&B)来记录我们的模型性能以及训练好的模型权重。为了运行我们的代码,如果您还没有账户,建议创建一个W&B账户(W&B)。所有代码均使用pytorch和pytorch-lightning编写。
运行实验
如上所述,每个笔记本代表一个实验。项目层次结构中的注释指明了图表或表格,以及实验所展示的具体论文。作为旁注,如果分配策略需要,您将需要训练相关的推理模型才能运行模拟实验(experiments/notebooks/wandb/simulation_tests.ipynb)。
训练新的模型(例如src/organsync/models/organsync_network.py)非常简单:
python -m experiments.models.organsync
(请运行python -m experiments.models.organsync --help以查看选项)。当训练完成后,模型将自动上传到W&B,以供实验中后续使用。*
引用
请按照以下方式引用我们的论文和/或代码:
@InProceedings{organsync,
title = {{Learning Queueing Policies for Organ Transplantation Allocation using Interpretable Counterfactual Survival Analysis}},
author = {Berrevoets, Jeroen and Alaa, Ahmed M. and Qian, Zhaozhi and Jordon, James and Gimson, Alexander E.S. and van der Schaar, Mihaela},
booktitle = {Proceedings of the 38th International Conference on Machine Learning},
pages = {792--802},
year = {2021},
editor = {Meila, Marina and Zhang, Tong},
volume = {139},
series = {Proceedings of Machine Learning Research},
month = {18--24 Jul},
publisher = {PMLR},
pdf = {http://proceedings.mlr.press/v139/berrevoets21a/berrevoets21a.pdf},
url = {http://proceedings.mlr.press/v139/berrevoets21a.html},
}
* 注意,我们重新训练了TransplantBenefit中使用的模型,以便与其他基准进行公平的比较,并在UNOS数据上进行比较。
项目详情
下载文件
下载适合您平台的文件。如果您不确定选择哪个,请了解更多关于安装包的信息。
源分布
构建分布
哈希 for organsync-0.1.5-py3-none-macosx_10_14_x86_64.whl
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | a844b43d57a5478a52a8361cd9a2b0e19fcc5413dccf8818963cf327fe929742 |
|
| MD5 | 1ab99273c1f25bb95af228215954a0cf |
|
| BLAKE2b-256 | f537cd13db1634db7f5ff0b0566ee9baeebfbc038e15402bece104039de1e8b8 |