为OpenMM轻松部署MD协议
项目描述




一个用于使用OpenMM启动分子动力学模拟的命令行应用程序
一些酷功能
无需编码 - 只需YAML输入文件!
- 对不同的输入文件格式提供智能支持
拓扑: PDB/PDBx, Mol2, Amber的PRMTOP, Charmm的PSF, Gromacs的TOP, Desmond的DMS
位置: PDB, COOR, INPCRD, CRD, GRO
速度: PDB, VEL
盒子向量: PDB, XSC, CSV, INPCRD, GRO
实现了回退方法,并尝试加载所有可能由ParmEd支持的其它内容。
选择您首选的轨迹格式(PDB,PDBx,DCD,HDF5,NETCDF,MDCRD)和检查点(Amber,CHARMM,OpenMM XML状态)。
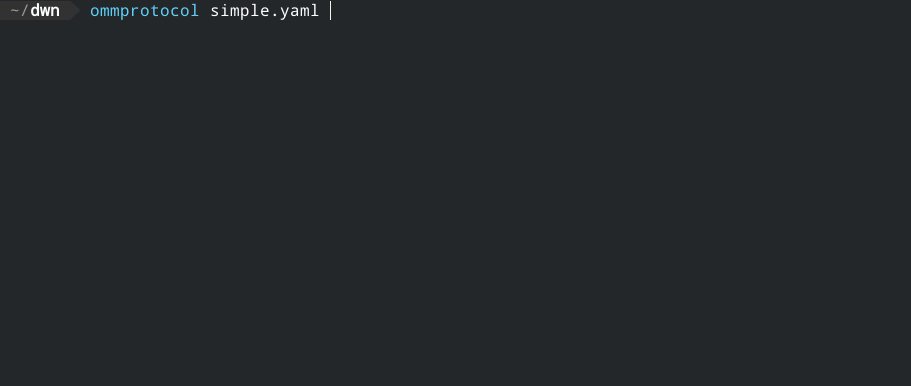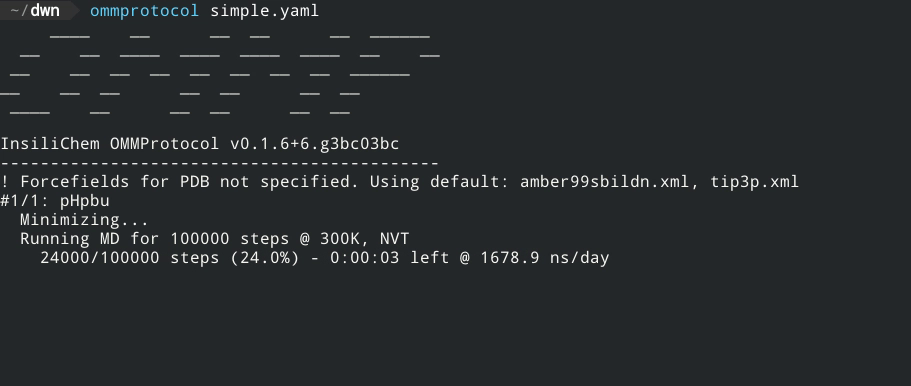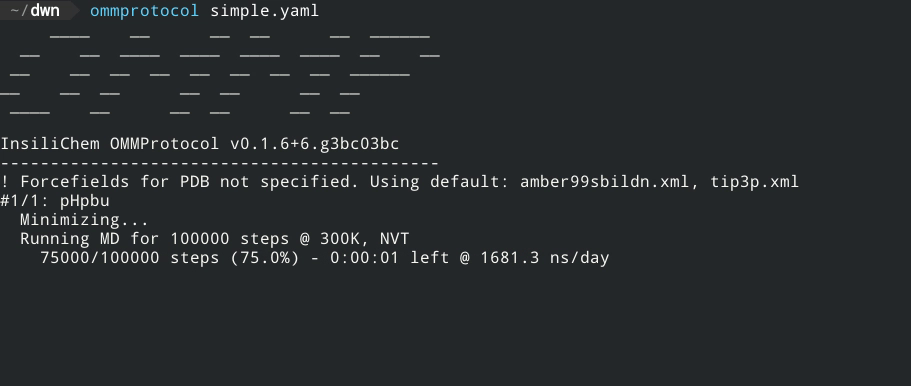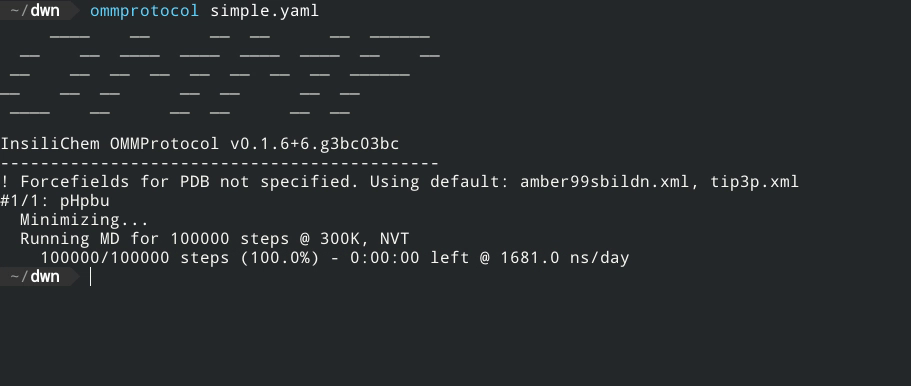
实时报告模拟进度,包括预估完成时间和速度。
每n步检查点。如果发生错误,还将创建紧急救援文件。
自动分割轨迹,便于处理。
安装与使用
下载最新安装程序,或者如果您已安装Anaconda/Miniconda,可以使用conda install -c omnia -c insilichem ommprotocol。更多详细信息在此。
安装后,您应该能够运行
ommprotocol <inputfile.yaml>
请查阅文档了解如何为OMMProtocol创建输入文件。
获取帮助

文档始终可在ReadTheDocs上找到。如果您在运行ommprotocol时遇到问题,请创建一个问题!同时,请确保访问我们的主页insilichem.com。
引用
OMMProtocol是科学软件,由公共研究资助(西班牙MINECO项目CTQ2014-54071-P,加泰罗尼亚政府项目2014SGR989和研究资助2015FI_B00768,COST行动CM1306)。如果您在科学出版物中使用OMMprotocol,请引用它。这将帮助我们衡量我们研究的影响和未来的资助!一篇论文正在准备中。在此期间,请引用此存储库URL。
@misc{ommprotocol,
author = {Jaime Rodríguez-Guerra Pedregal and
Lur Alonso-Cotchico and
Lorea Velasco and
Jean-Didier Maréchal},
title = {OMMProtocol: A command line application to launch molecular dynamics simulations with OpenMM},
url = {https://github.com/insilichem/ommprotocol}
}项目详情
ommprotocol-0.1.11.tar.gz的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 9c69eb511c36fd21ab77f8cde8431d1af5d3d5f4b3b883aadfce93e1245d3e30 |
|
| MD5 | 3e5c4c94c33c9dcad80c306449503fb3 |
|
| BLAKE2b-256 | 1f7b3ce81741d568012e4e1333dfd47dd84268c6bbb11e813b0c376b8fa1addf |



