MAPR是OMERO.web的Python插件
项目描述


MAPR
OMERO.mapr 是一个 OMERO.web 应用,允许通过 Map 注释的形式浏览与图像关联的属性。
它被图像数据资源(Image Data Resource)广泛使用,允许用户通过基因、表型、生物体等各种类别查找数据。
在 OMERO 中,Map 注释是一系列命名属性或“键值对”,可以用于注释多种类型的数据。注释可以分配一个 命名空间 来指明注释的来源和目的。
通过 Insight 客户端或 webclient 创建的 Map 注释都拥有命名空间 openmicroscopy.org/omero/client/mapAnnotation,而通过 OMERO API 由其他工具创建的其他 Map 注释应拥有自己独特的命名空间。
我们可以配置 OMERO.mapr 以搜索特定 命名空间 的 Map 注释,在指定的 键 下查找 值。例如,在键 Gene Symbol 或 Gene Identifier 和命名空间 openmicroscopy.org/mapr/gene 下搜索值。
需求
OMERO.web 5.6 或更高版本。
从 PyPI 安装
本节假设 OMERO.web 已安装。注意:配置设置(见下文)是必需的,否则应用无法工作。
使用 pip 安装应用
$ pip install omero-mapr
将应用添加到已安装应用列表中
$ omero config append omero.web.apps '"omero_mapr"'
配置设置
您需要配置用户可以搜索的命名空间和键。
用户编辑的 Map 注释
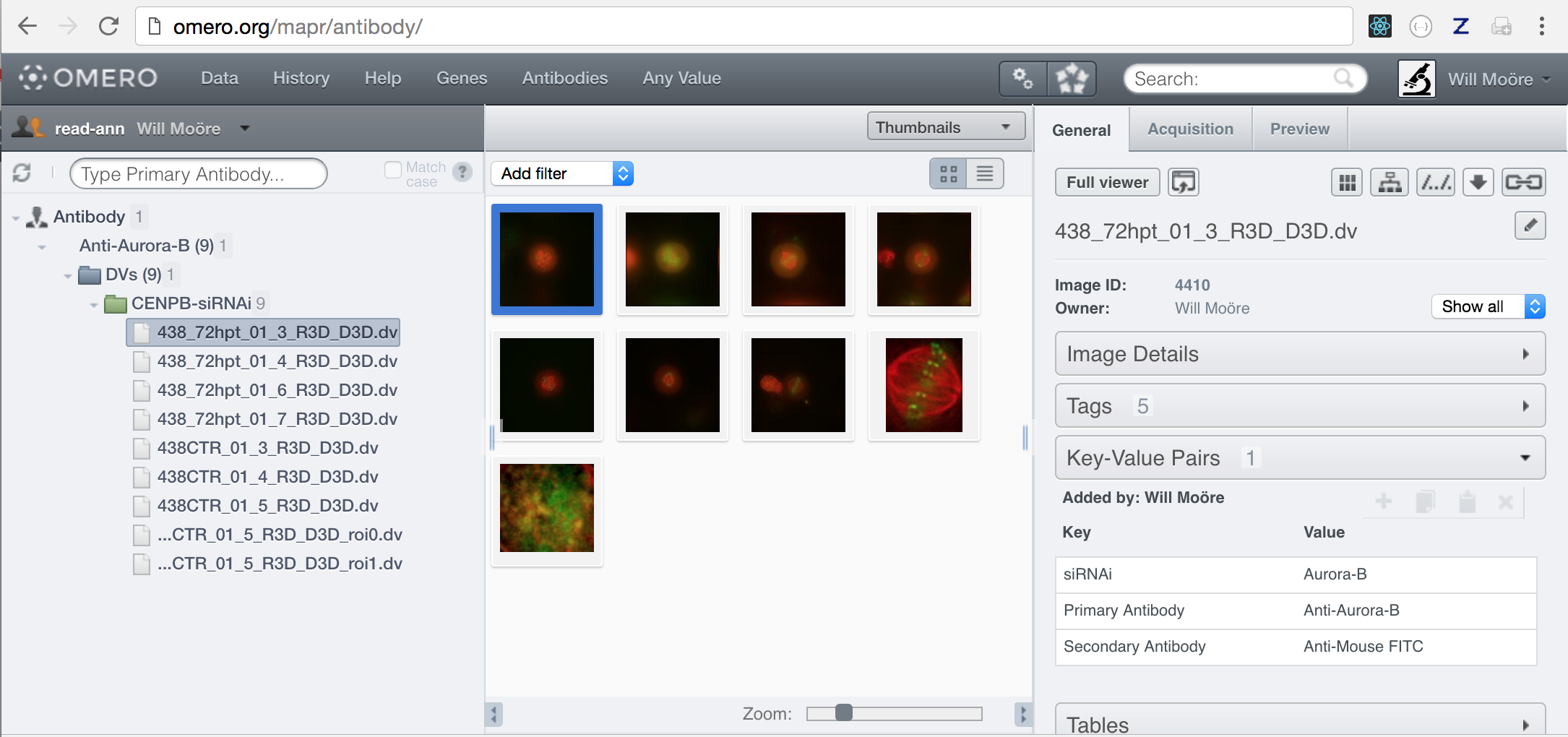
在键值面板中使用 webclient 或 Insight 添加的 Map 注释使用特定的“客户端”命名空间。因此,我们可以配置 OMERO.mapr 使用命名空间 openmicroscopy.org/omero/client/mapAnnotation 搜索这些注释。例如,为了搜索“主要抗体”或“二级抗体”的值,我们可以添加
$ omero config append omero.web.mapr.config '{"menu": "antibody", "config":{"default":["Primary Antibody"], "all":["Primary Antibody", "Secondary Antibody"], "ns":["openmicroscopy.org/omero/client/mapAnnotation"], "label":"Antibody"}}'
我们可以在 webclient 页面的顶部添加一个“抗体”链接,带有一个工具提示“查找抗体值”。viewname 应采用 maprindex_{menu} 的形式,其中 {menu} 是前一个配置中的 menu 值。
$ omero config append omero.web.ui.top_links '["Antibodies", {"viewname": "maprindex_antibody"}, {"title": "Find Antibody values"}]'
重启 web 后,我们现在可以搜索抗体
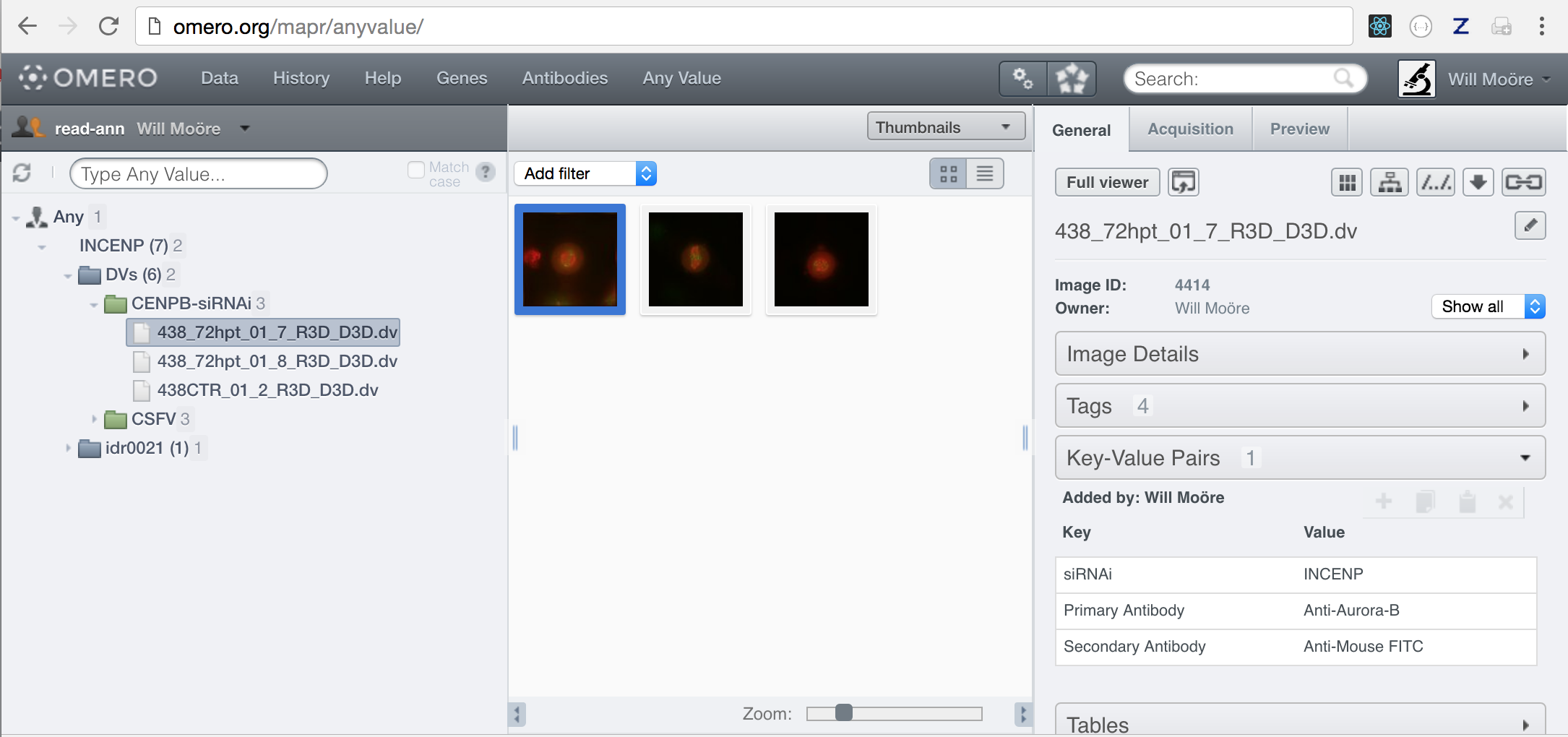
我们还可以指定一个空的键列表来搜索 任何 值。
$ omero config append omero.web.mapr.config '{"menu": "anyvalue", "config":{"default":["Any Value"], "all":[], "ns":["openmicroscopy.org/omero/client/mapAnnotation"], "label":"Any"}}'
# Top link
$ omero config append omero.web.ui.top_links '["Any Value", {"viewname": "maprindex_anyvalue"}, {"title": "Find Any Value"}]'
重启 web 后,我们现在可以搜索任何值,例如“INCENP”
其他 Map 注释
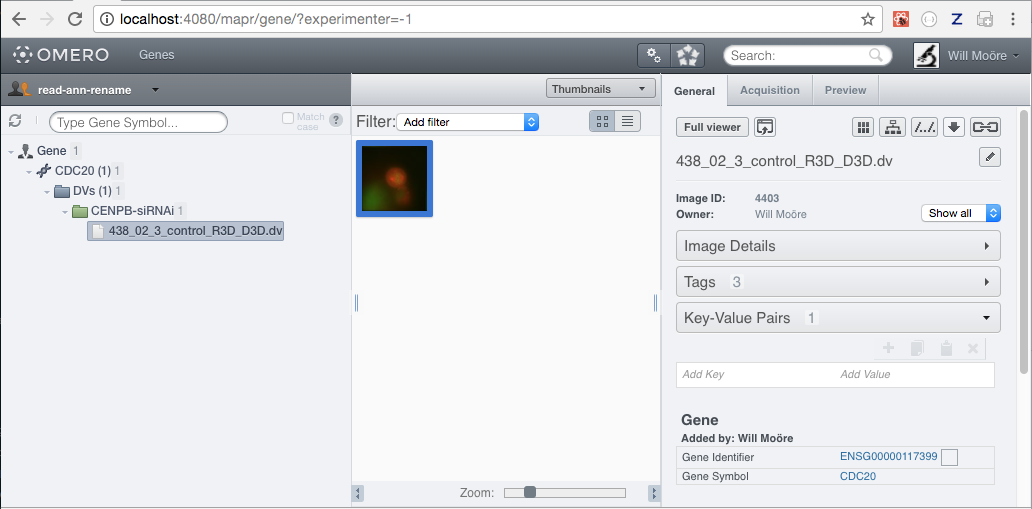
在这个例子中,我们想搜索命名空间 openmicroscopy.org/mapr/gene 的 Map 注释,在 Gene Symbol 和 Gene Identifier 键下搜索属性。
$ omero config append omero.web.mapr.config '{"menu": "gene","config": {"default": ["Gene Symbol"],"all": ["Gene Symbol", "Gene Identifier"],"ns": ["openmicroscopy.org/mapr/gene"],"label": "Gene"}}'
现在添加一个顶部链接 Genes,带有一个工具提示 Find Gene annotations,这将带我们到 gene 搜索页面。将 query_string 参数添加到 URL 中,"experimenter": -1 指定我们要搜索所有用户的。
$ omero config append omero.web.ui.top_links '["Genes", {"viewname": "maprindex_gene", "query_string": {"experimenter": -1}}, {"title": "Find Gene annotations"}]'
最后,我们可以在位于 Screen -> Plate -> Well 或 Project -> Dataset -> Image 层次结构中的 Image 上添加一个 map 注释。此代码使用 OMERO Python API 添加与上述配置相对应的 map 注释。
key_value_data = [["Gene Identifier","ENSG00000117399"],
["Gene Identifier URL", "http://www.ensembl.org/id/ENSG00000117399"],
["Gene Symbol","CDC20"]]
map_ann = omero.gateway.MapAnnotationWrapper(conn)
map_ann.setValue(key_value_data)
map_ann.setNs("openmicroscopy.org/mapr/gene")
map_ann.save()
image = conn.getObject('Image', 2917)
image.linkAnnotation(map_ann)
现在以正常方式重新启动OMERO.web,以使上述配置生效。您现在应该能够浏览到基因页面,并搜索CDC20或ENSG00000117399。
外部URL Favicon
Mapr可以自动将URL转换为favicon链接。要使用此功能,键(如基因标识符)必须在配置的“所有”列表中,如上图所示,并且必须跟随着名为基因标识符URL的键值对。一个链接到外部URL的favicon将被附加到基因标识符行中,并且基因标识符URL键值对将被隐藏。OMERO.web必须配置Django redis缓存https://docs.openmicroscopy.org/omero/5/sysadmins/unix/install-web/walkthrough/omeroweb-install-centos7-ice3.6.html?highlight=redis#configuring-omero-web,该缓存用于缓存通过Google服务获得的favicon。
测试
测试MAPR需要OMERO.server运行。运行测试(包括自包含的OMERO.server,需要docker)
docker-compose -f docker/docker-compose.yml up --build --abort-on-container-exit docker-compose -f docker/docker-compose.yml rm -fv
许可证
MAPR遵循AGPL发布。
版权
2016-2021,Open Microscopy Environment
项目详情
下载文件
下载适用于您的平台的文件。如果您不确定选择哪一个,请了解有关安装包的更多信息。
源分布
构建分布
omero-mapr-0.5.2.tar.gz的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 721dede883d96dd5b9dd2b2b2c8f0c5492ab3688aef47a8979542f903836675f |
|
| MD5 | 0d95ebb41a0766cd641655ede427bd40 |
|
| BLAKE2b-256 | 5a43e0f8f2eee2a982492e0790ec6b05a689fb8765ac056b416d26f0497ce478 |







