用于本体论的NetworkX
项目描述
基于NetworkX的Python库,用于表示本体
摘要
nxontology是一个Python库,使用NetworkX图来表示本体。目前,主要的功能领域是计算节点对之间的相似度度量。
使用方法
在此,我们将使用金属本体的例子
注意,NXOntology将本体表示为networkx.DiGraph,其中边方向从超项到子项。
给定一个NXOntology实例,以下是计算内在相似度指标的方法。
from nxontology.examples import create_metal_nxo
metals = create_metal_nxo()
# Freezing the ontology prevents adding or removing nodes or edges.
# Frozen ontologies cache expensive computations.
metals.freeze()
# Get object for computing similarity, using the Sanchez et al metric for information content.
similarity = metals.similarity("gold", "silver", ic_metric="intrinsic_ic_sanchez")
# Access a single similarity metric
similarity.lin
# Access all similarity metrics
similarity.results()
最后一行输出一个类似以下字典的字典
{
'node_0': 'gold',
'node_1': 'silver',
'node_0_subsumes_1': False,
'node_1_subsumes_0': False,
'n_common_ancestors': 3,
'n_union_ancestors': 5,
'batet': 0.6,
'batet_log': 0.5693234419266069,
'ic_metric': 'intrinsic_ic_sanchez',
'mica': 'coinage',
'resnik': 0.8754687373538999,
'resnik_scaled': 0.48860840553061435,
'lin': 0.5581154235118403,
'jiang': 0.41905978419640516,
'jiang_seco': 0.6131471927654584,
}
还可以将两个节点之间的相似度可视化
from nxontology.viz import create_similarity_graphviz
gviz = create_similarity_graphviz(
# similarity instance from above
similarity,
# show all nodes (defaults to union of ancestors)
nodes=list(metals.graph),
)
# draw to PNG file
gviz.draw("metals-sim-gold-silver-all.png"))
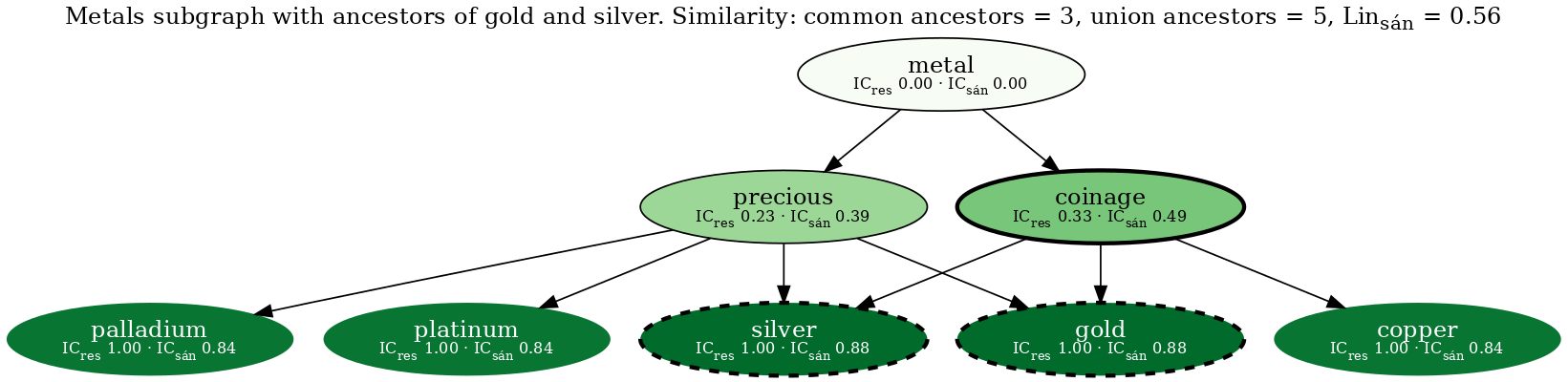
结果如下所示
两个查询节点(黄金和白银)用粗虚线勾勒。节点填充颜色与Sánchez信息内容相对应,因此颜色越深的节点具有越高的IC。最有信息量的共同祖先(铸币)用粗实线勾勒。不是黄金或白银祖先的节点没有可见轮廓。
加载本体
Pronto支持从以下文件格式读取本体
- 开放生物医学本体1.4:使用
.obo扩展名,采用fastobo解析器。 - OBO Graphs JSON:使用
.json扩展名,采用fastobo解析器。 - 本体网络语言2 RDF/XML:使用
.owl扩展名,采用pronto的RdfXMLParser。
文件可以是本地的或网络位置(以https、http或ftp开始的URL)。Pronto可以检测并处理gzip、bzip2和xz压缩。
以下是使用pronto加载本体的Gene Ontology操作的示例
>>> from nxontology.imports import from_file
>>> # versioned URL for the Gene Ontology
>>> url = "http://release.geneontology.org/2021-02-01/ontology/go-basic.json.gz"
>>> nxo = from_file(url)
>>> nxo.n_nodes
44085
>>> # similarity between "myelination" and "neurogenesis"
>>> sim = nxo.similarity("GO:0042552", "GO:0022008")
>>> round(sim.lin, 2)
0.21
>>> import networkx as nx
>>> # Gene Ontology domains are disconnected, expect 3 components
>>> nx.number_weakly_connected_components(nxo.graph)
3
>>> # Note however that the default from_file reader only uses "is a" relationships.
>>> # We can preserve all GO relationship types as follows
>>> from collections import Counter
>>> import pronto
>>> from nxontology import NXOntology
>>> from nxontology.imports import pronto_to_multidigraph, multidigraph_to_digraph
>>> go_pronto = pronto.Ontology(handle=url)
>>> go_multidigraph = pronto_to_multidigraph(go_pronto)
>>> Counter(key for _, _, key in go_multidigraph.edges(keys=True))
Counter({'is a': 71509,
'part of': 7187,
'regulates': 3216,
'negatively regulates': 2768,
'positively regulates': 2756})
>>> go_digraph = multidigraph_to_digraph(go_multidigraph, reduce=True)
>>> go_nxo = NXOntology(go_digraph)
>>> # Notice the similarity increases due to the full set of edges
>>> round(go_nxo.similarity("GO:0042552", "GO:0022008").lin, 3)
0.699
>>> # Note that there is also a dedicated reader for the Gene Ontology
>>> from nxontology.imports import read_gene_ontology
>>> read_gene_ontology(release="2021-02-01")
用户也可以创建自己的networkx.DiGraph来使用此包。
预构建本体
nxontology-data存储库为许多流行的本体/分类法创建了NXOntology对象。
安装
nxontology可以通过从PyPI使用pip安装
# standard installation
pip install nxontology
# installation with viz extras
pip install nxontology[viz]
需要额外的viz依赖项才能使用nxontology.viz模块。这包括pygraphviz,它需要一个预先存在的graphviz安装。
开发
一些有用的开发命令
# create a virtual environment for development
python3 -m venv .venv
# activate virtual environment
source .venv/bin/activate
# install package for development
pip install --editable ".[dev,viz]"
# Set up the git pre-commit hooks.
# `git commit` will now trigger automatic checks including linting.
pre-commit install
# Run all pre-commit checks (CI will also run this).
pre-commit run --all
# run tests
pytest
在GitHub上创建发行版。由release.yaml定义的发行操作将构建发行版并将其上传到PyPI。软件包版本由setuptools_scm自动从git标签生成。
参考书目
以下是一些具有计算本体语义相似度度量的代码的替代项目列表
- 在sharispe/slib的Java中,位于语义度量库 & 工具包。
- 在lasigeBioTM/DiShIn的Python中,位于DiShIn。
- 在gsi-upm/sematch的Python中,位于Sematch。
- 在cran/ontologySimilarity中镜像的ontologySimilarity。是R包ontologyX套件的一部分。
- 在bio-ontology-research-group/machine-learning-with-ontologies(汇编)中,关于使用本体的机器学习材料
以下是关于从本体派生的相似度度量的参考文献列表。请随意添加任何提供对支持此包的算法有用的上下文和详细信息的参考文献。可以像manubot cite --yml doi:10.1016/j.jbi.2011.03.013那样生成参考文献的元数据。将CSL YAML输出添加到media/bibliography.yaml中将缓存元数据,并允许在发生错误时手动编辑。
-
生物医学本体的语义相似性
Catia Pesquita,Daniel Faria,André O. Falcão,Phillip Lord,Francisco M. Couto
PLoS Computational Biology (2009-07-31) https://doi.org/cx8h87
DOI: 10.1371/journal.pcbi.1000443 · PMID: 19649320 · PMCID: PMC2712090 -
WordNet语义相似性内在信息含量度量。
Nuno Seco, Tony Veale, Jer Hayes
第16届欧洲人工智能会议(ECAI-04)论文集,(2004) https://citeseerx.ist.psu.edu/viewdoc/summary?doi=10.1.1.1065.1695 -
基于GO的蛋白质语义相似性度量:系统评估
Catia Pesquita, Daniel Faria, Hugo Bastos, António EN Ferreira, André O Falcão, Francisco M Couto
BMC生物信息学 (2008-04-29) https://doi.org/cmcgw6
DOI: 10.1186/1471-2105-9-s5-s4 · PMID: 18460186 · PMCID: PMC2367622 -
本体语义相似性与机器学习
Maxat Kulmanov, Fatima Zohra Smaili, Xin Gao, Robert Hoehndorf
生物信息学简报 (2020-10-13) https://doi.org/ghfqkt
DOI: 10.1093/bib/bbaa199 · PMID: 33049044 -
分类学中的语义相似性:基于信息的方法及其在自然语言歧义问题中的应用
P. Resnik
人工智能研究杂志 (1999-07-01) https://doi.org/gftcpz
DOI: 10.1613/jair.514 -
相似性信息论定义
Dekang Lin
ICML (1998) https://api.semanticscholar.org/CorpusID:5659557 -
ontologyX:一套用于处理本体数据的R包
Daniel Greene, Sylvia Richardson, Ernest Turro
生物信息学 (2017-01-05) https://doi.org/f9k7sx
DOI: 10.1093/bioinformatics/btw763 · PMID: 28062448 · PMCID: PMC5386138 -
用于测量本体中语义相似性的内在信息含量度量
Md. Hanif Seddiqui, Masaki Aono
第7届亚太地区概念建模会议论文集 - 第110卷 (2010-01-01) https://dl.acm.org/doi/10.5555/1862330.1862343
ISBN: 9781920682927 -
本体概念之间的析取共享信息:应用于基因本体
Francisco M Couto, Mário J Silva
生物医学语义学杂志 (2011) https://doi.org/fnb73v
DOI: 10.1186/2041-1480-2-5 · PMID: 21884591 · PMCID: PMC3200982 -
统一基于本体语义相似性度量的框架:生物医学领域的研究
Sébastien Harispe, David Sánchez, Sylvie Ranwez, Stefan Janaqi, Jacky Montmain
生物医学信息学杂志 (2014-04) https://doi.org/f52557
DOI: 10.1016/j.jbi.2013.11.006 · PMID: 24269894 -
化学信息学中的语义相似性
João D. Ferreira, Francisco M. Couto
IntechOpen (2020-07-15) https://doi.org/ghh2d4
DOI: 10.5772/intechopen.89032 -
计算生物医学中语义相似性的本体度量
Montserrat Batet, David Sánchez, Aida Valls
生物医学信息学杂志 (2011-02) https://doi.org/dfhkjv
DOI: 10.1016/j.jbi.2010.09.002 · PMID: 20837160 -
生物医学领域的语义相似性:跨知识源的评价
维贾伊·N·加拉,辛西娅·布兰特
BMC生物信息学 (2012-10-10) https://doi.org/gb8vpn
DOI: 10.1186/1471-2105-13-261 · PMID: 23046094 · PMCID: PMC3533586 -
生物医学领域的语义相似性估计:基于本体论的信息论视角
大卫·桑切斯,蒙托萨·巴特
生物医学信息学杂志 (2011-10) https://doi.org/d2436q
DOI: 10.1016/j.jbi.2011.03.013 -
基于本体论的信息内容计算
大卫·桑切斯,蒙托萨·巴特,大卫·伊塞恩
基于知识的系统 (2011-03) https://doi.org/cwzw4r
DOI: 10.1016/j.knosys.2010.10.001 -
利用同义词和多义词提高基于内在信息内容的语义相似性评估
蒙托萨·巴特,大卫·桑切斯
人工智能评论 (2019-06-03) https://doi.org/ghnfmt
DOI: 10.1007/s10462-019-09725-4 -
考虑本体概念的不相交共同子集的基于内在信息内容的语义相似性度量
阿比吉特·阿迪卡里,比斯瓦纳特·杜塔,阿尼梅什·杜塔,迪普吉约蒂·莫达尔,希旺·辛格
信息科学与技术协会杂志 (2018-08) https://doi.org/gd2j5b
DOI: 10.1002/asi.24021
项目详情
下载文件
下载适用于您平台的文件。如果您不确定选择哪个,请了解更多关于安装包的信息。
源代码分发
构建分发
nxontology-0.5.0.tar.gz的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 727712c2bdbcaf7113f0012aa66bb9097ddef375f604939fb279b0871aed2c2f |
|
| MD5 | 72c6a8d6b594fd86bccbdd41d998e968 |
|
| BLAKE2b-256 | d544a5f52f698a580982b3afc53f6c14edb64d27acc07e78ad12aa1d9ee8be6e |
nxontology-0.5.0-py3-none-any.whl的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | c95d2d47c2ce8c0bb48e839a3bdc70e489c13f4579b6e88b8d93a2be87c2c07f |
|
| MD5 | b6321c129b7338146a4cee09f0c30971 |
|
| BLAKE2b-256 | 00c0a7e608c1e86c4164468e6e969e330a5c47f0f7393ef7dabdfc91cc8b36cd |