用于开发3D图像处理神经网络模型的框架。
项目描述
显而易见
显而易见 是一个用于3D图像处理的深度学习框架。它实现了从最近文献中提取的几个3D卷积模型,可用于与任何TensorFlow或Keras模型一起使用的体素数据加载和增强方法,3D数据的损失和度量,以及用于模型训练、评估、预测和迁移学习的简单工具。
无脑器 还提供了用于脑提取、脑分割、脑生成等任务的预训练模型。请参阅 训练 模型 存储库以获取更多信息。
无脑器 项目由NIH RF1MH121885支持,并采用Apache 2.0许可证进行分发。它是在NIH R01 EB020470的支持下开始的。
目录
实现
模型
| 模型 | 类型 | 应用 |
|---|---|---|
| Highresnet (source) | 监督 | 分割/分类 |
| Unet (source) | 监督 | 分割/分类 |
| Vnet (source) | 监督 | 分割/分类 |
| Meshnet (source) | 监督 | 分割/分类 |
| 贝叶斯 Meshnet (source) | 贝叶斯监督 | 分割/分类 |
| 贝叶斯 Vnet | 贝叶斯监督 | 分割/分类 |
| 半贝叶斯 Vnet | 半贝叶斯监督 | 分割/分类 |
| DCGAN | 自监督 | 生成模型 |
| 渐进式 GAN | 自监督 | 生成模型 |
| 3D 自动编码器 | 自监督 | 知识表示/降维 |
| 3D 渐进式自动编码器 | 自监督 | 知识表示/降维 |
| 3D SimSiam (source) | 自监督 | 孪生表示学习 |
Dropout和正则化层
伯努利dropout层,康克雷迪dropout层,高斯dropout,分组归一化层,自定义填充层
损失
Dice,Jaccard,Tversky,ELBO,Wasserstien,梯度惩罚
指标
Dice,广义Dice,Jaccard,Hamming,Tversky
增强方法
空间变换
中心裁剪,空间常数填充,随机裁剪,调整大小,随机翻转(左右)
强度变换
添加高斯噪声,最小-最大强度缩放,自定义强度缩放,强度掩码,对比度调整
仿射变换
包括旋转、平移、反射的仿射变换。
指南Jupyter笔记本 
请参考指南目录中的 Jupyter 笔记本以开始使用 Nobrainer,有关指南请参阅 此处。在 Google Colaboratory 中 尝试它们!
安装
容器
我们建议使用官方的 Nobrainer Docker 容器,它包含使用该框架所需的所有依赖项。请查看 DockerHub 上的可用镜像 此处
GPU支持
支持 GPU 的 Nobrainer 容器使用 Tensorflow jupyter GPU 容器。请检查容器中安装的 CUDA 版本。容器中不包含 Nvidia 驱动程序。
$ docker pull neuronets/nobrainer:latest-gpu
$ singularity pull docker://neuronets/nobrainer:latest-gpu
仅CPU
此容器可用于所有具有 Docker 或 Singularity 的系统,并且不需要特殊硬件。然而,此容器不应用于模型训练(这将非常慢)。
$ docker pull neuronets/nobrainer:latest-cpu
$ singularity pull docker://neuronets/nobrainer:latest-cpu
pip
Nobrainer 也可以使用 pip 进行安装。
$ pip install nobrainer
使用预训练网络
预训练网络可在 训练模型 存储库中获得。可以使用 nobrainer predict 命令行工具或 Python 进行预测。类似地,可以使用 nobrainer generate 命令行工具或 Python 进行生成。
预测T1加权脑扫描的脑掩码
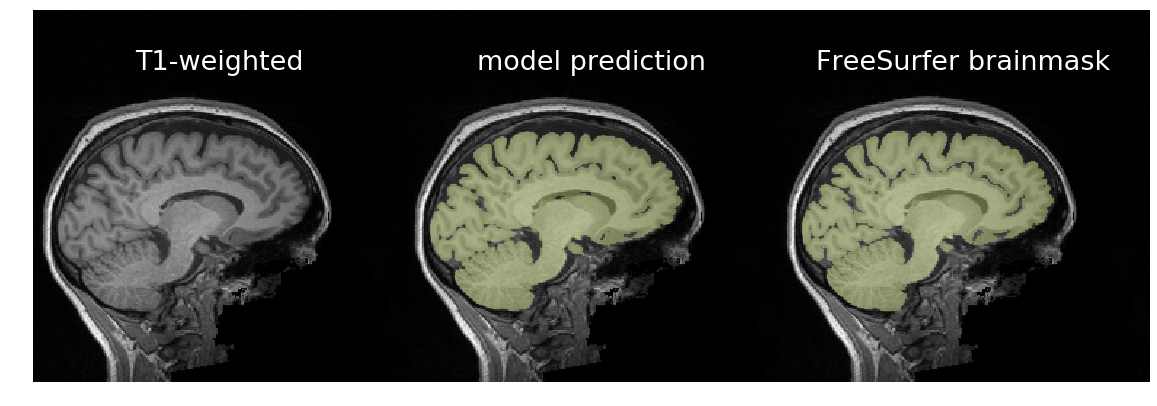
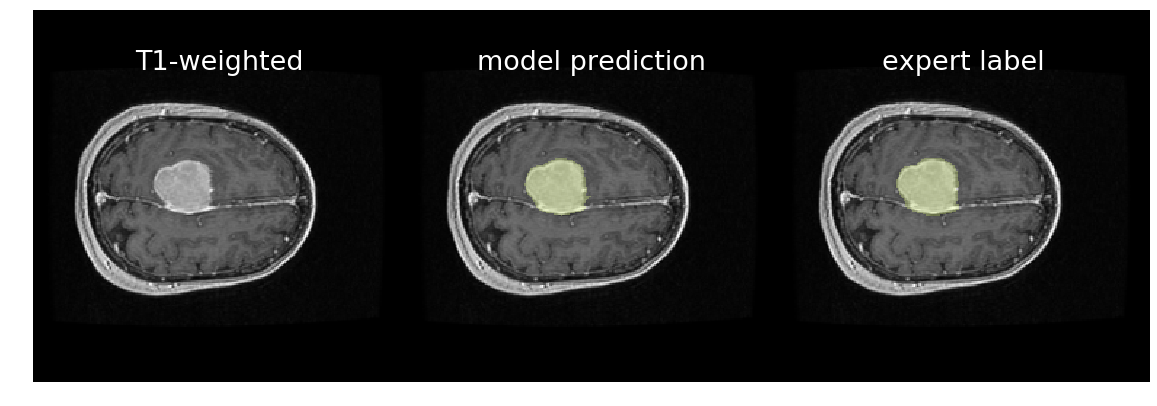
在以下示例中,我们将使用一个用于脑提取并记录在 训练模型 中的 3D U-Net。
在基本情况下,我们将 T1w 扫描通过模型进行预测。
# Get sample T1w scan.
wget -nc https://dl.dropbox.com/s/g1vn5p3grifro4d/T1w.nii.gz
docker run --rm -v $PWD:/data neuronets/nobrainer \
predict \
--model=/models/neuronets/brainy/0.1.0/brain-extraction-unet-128iso-model.h5 \
--verbose \
/data/T1w.nii.gz \
/data/brainmask.nii.gz
对于期望一个预测区域的二值分割(例如脑提取),我们可以通过移除所有未连接到最大连续标签的预测来减少假阳性。
# Get sample T1w scan.
wget -nc https://dl.dropbox.com/s/g1vn5p3grifro4d/T1w.nii.gz
docker run --rm -v $PWD:/data neuronets/nobrainer \
predict \
--model=/models/neuronets/brainy/0.1.0/brain-extraction-unet-128iso-model.h5 \
--largest-label \
--verbose \
/data/T1w.nii.gz \
/data/brainmask-largestlabel.nii.gz
由于网络是在随机旋转的数据上训练的,它应该对方向无感知。因此,我们可以旋转体积,对其预测,然后在预测中撤销旋转,并将原始体积的预测与它平均。这可以提高整体预测的质量,但至少会加倍处理时间。要启用此功能,请在 nobrainer predict 中使用 --rotate-and-predict 标志。
# Get sample T1w scan.
wget -nc https://dl.dropbox.com/s/g1vn5p3grifro4d/T1w.nii.gz
docker run --rm -v $PWD:/data neuronets/nobrainer \
predict \
--model=/models/neuronets/brainy/0.1.0/brain-extraction-unet-128iso-model.h5 \
--rotate-and-predict \
--verbose \
/data/T1w.nii.gz \
/data/brainmask-withrotation.nii.gz
结合上述内容,我们通常可以通过在 nobrainer predict 中同时使用 --rotate-and-predict 和 --largest-label 来实现最佳的脑提取。
# Get sample T1w scan.
wget -nc https://dl.dropbox.com/s/g1vn5p3grifro4d/T1w.nii.gz
docker run --rm -v $PWD:/data neuronets/nobrainer \
predict \
--model=/models/neuronets/brainy/0.1.0/brain-extraction-unet-128iso-model.h5 \
--largest-label \
--rotate-and-predict \
--verbose \
/data/T1w.nii.gz \
/data/brainmask-maybebest.nii.gz
生成合成T1加权脑扫描
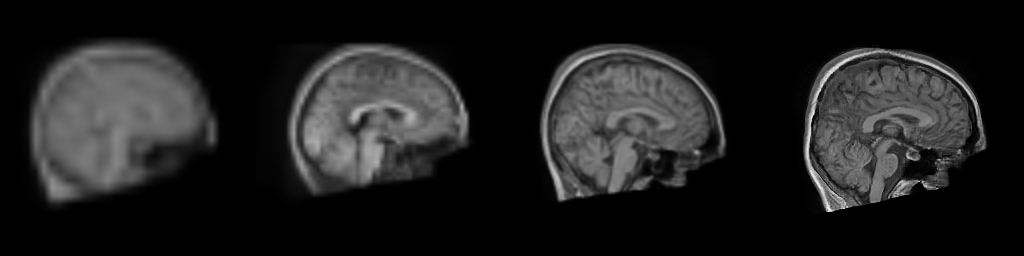
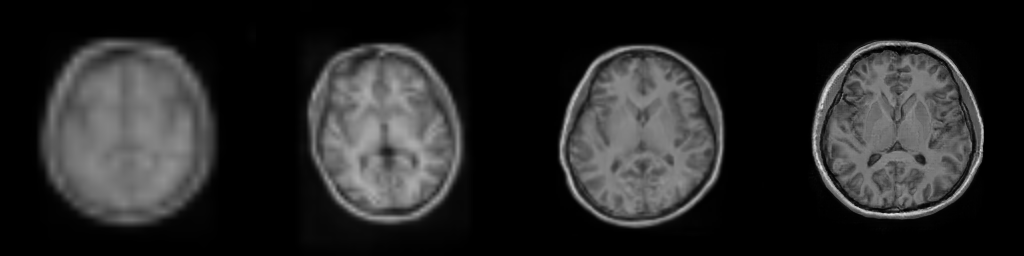
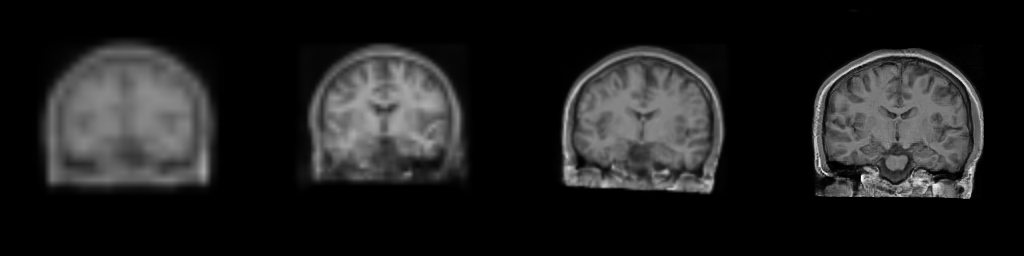
在以下示例中,我们将使用为脑图像生成训练的渐进生成对抗网络,并在训练模型中进行说明。
在基本情况下,我们通过模型为给定分辨率生成T1w扫描。我们需要传递包含在训练网络时创建的模型目录(tf.SavedModel)。
docker run --rm -v $PWD:/data neuronets/nobrainer \
generate \
--model=/models/neuronets/braingen/0.1.0 \
--output-shape=128 128 128 \
/data/generated.nii.gz
我们还可以使用相同的潜在变量生成脑图像的多个分辨率,以可视化进程。
# Get sample T1w scan.
docker run --rm -v $PWD:/data neuronets/nobrainer \
generate \
--model=/models/neuronets/braingen/0.1.0 \
--multi-resolution \
/data/generated.nii.gz
在上面的示例中,多分辨率图像将保存为generated_res_{resolution}.nii.gz
迁移学习
预训练模型可用于迁移学习。为了避免在预训练模型中忘记重要信息,您可以对内核权重应用正则化,并使用低学习率。有关更多信息,请参阅Nobrainer迁移学习指南笔记本。
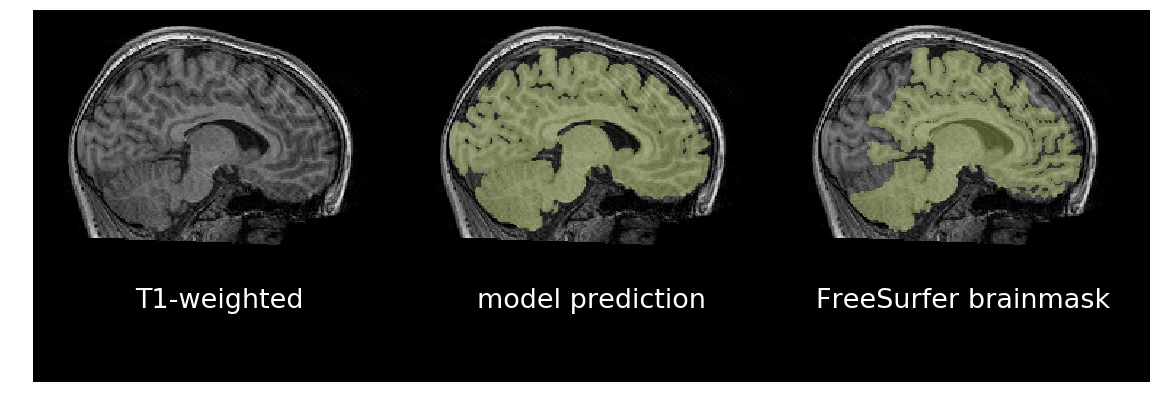
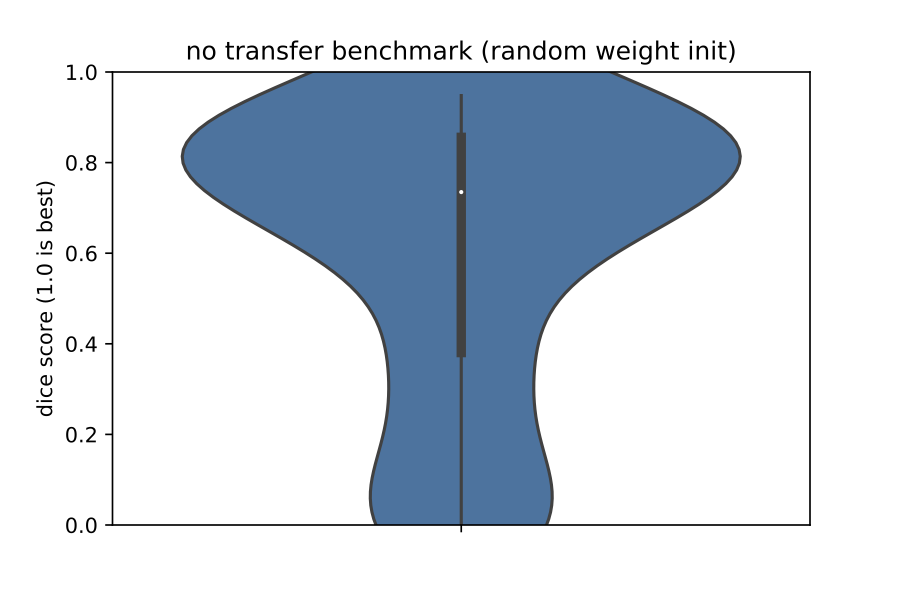
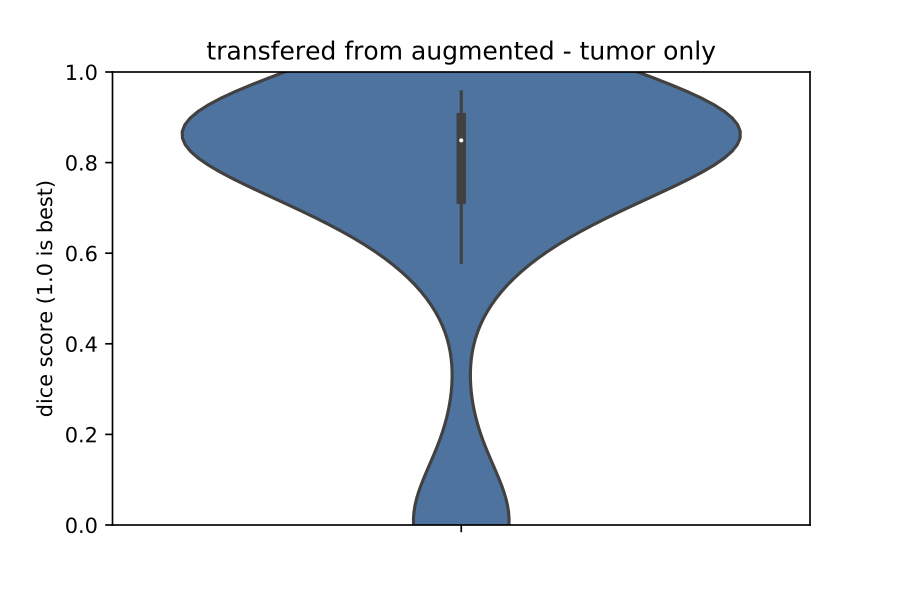
作为迁移学习的示例,@kaczmarj重新训练了一个脑提取模型,用于标记3D T1加权、对比增强MRI扫描中的脑膜瘤。原始模型是公开的,并在来自健康参与者的10,000个T1加权MRI脑扫描上进行训练。这些扫描都是研究扫描(即非临床),且没有使用任何对比剂。另一方面,脑膜瘤数据集由相对较少的扫描组成,所有扫描都是临床的,并使用了钆作为对比剂。您可以在下面观察对比差异。

尽管两个数据集之间存在差异,但迁移学习导致模型比从随机初始化的权重训练的模型要好得多。作为证据,请参阅以下验证集上Dice系数的提琴图。在左图是使用从随机初始化的权重训练的模型获得的预测Dice系数,而在右图是使用迁移学习模型获得的预测Dice系数。一般来说,右边的Dice系数更高,Dice分数的方差更低。总的来说,右边的模型比左边的模型更准确、更健壮。
包布局
nobrainer.io:输入/输出方法nobrainer.layers:自定义层,符合Keras APInobrainer.losses:体积分割的损失函数nobrainer.metrics:体积分割的度量nobrainer.models:预定义的Keras模型nobrainer.training:训练实用工具(支持在单个和多个GPU上训练)nobrainer.transform:数据增强的随机刚性变换nobrainer.volume:创建tf.data.Dataset和数据增强实用工具
引用
如果您使用此包,请引用它。
问题或问题
如果您对Nobrainer有任何疑问或在使用框架时遇到任何问题,请提交GitHub问题。
项目详情
下载文件
下载适用于您平台的文件。如果您不确定选择哪个,请了解更多关于安装包的信息。
源代码分发
构建分发
nobrainer-1.2.1.tar.gz 的散列
| 算法 | 散列摘要 | |
|---|---|---|
| SHA256 | fbad00b459733ff86ef75dd46b603336183e4cbfeb13248a41db251bb3b3b11f |
|
| MD5 | 464a7eb1216e2368a129f17f97e9b6a7 |
|
| BLAKE2b-256 | fd6e602906691c7f12cdd822cf2d6de53f94726ed3f7b1015667e7a60d6c18ab |
nobrainer-1.2.1-py3-none-any.whl 的散列
| 算法 | 散列摘要 | |
|---|---|---|
| SHA256 | d31759dcddce43fcefa3edb878db6c49a9e957ab97864d51a0bf74d6e630d283 |
|
| MD5 | d9ef7b0804dfcd0bfe865af44b6c26c1 |
|
| BLAKE2b-256 | f4bd9ef30516e4c58c1e2e3070661bd14f6c92c1c01e518170b45d0c5bdda1e8 |