从VCF预测剪切变异效应
项目描述
MMSplice & MTSplice
从VCF预测(组织特异性)剪切变异效应。MTSplice与MMSplice集成,具有相同的API。
论文:Cheng等人 https://doi.org/10.1101/438986,https://www.biorxiv.org/content/10.1101/2020.06.07.138453v1
安装
外部依赖
pip install cyvcf2 cython
推荐使用Conda安装
conda install cyvcf2 cython -y
pip install mmsplice
在线运行MMSplice
您可以使用以下google colab笔记本在线运行mmsplice
准备
1. 准备注释(gtf)文件
可以从ensembl或gencode下载GTF格式的标准人类基因注释文件。《MMSplice》可以直接使用这些文件,但是强烈建议进行一些过滤。
- 过滤蛋白质编码基因。
2. 准备变异(VCF)文件
一个正确格式的VCF文件可以与《MMSplice》一起使用,但是以下步骤将降低假阳性的可能性
- 质量过滤。低质量变异会导致不可靠的预测。
- 避免在一行中呈现多个变异,可以将它们拆分成多行。执行此操作的示例代码
bcftools norm -m-both -o out.vcf in.vcf.gz
- 左归一化。例如,GGCA-->GG 不是左归一化的,而 GCA-->G 是。关于遗传变异的统一表示细节,请参阅 Tan 等人。
bcftools norm -f reference.fasta -o out.vcf in.vcf
3. 准备参考基因组(fasta)文件
人类参考fasta文件可以从ensembl/gencode下载。请确保染色体名称与您使用的GTF注释文件匹配。
示例代码
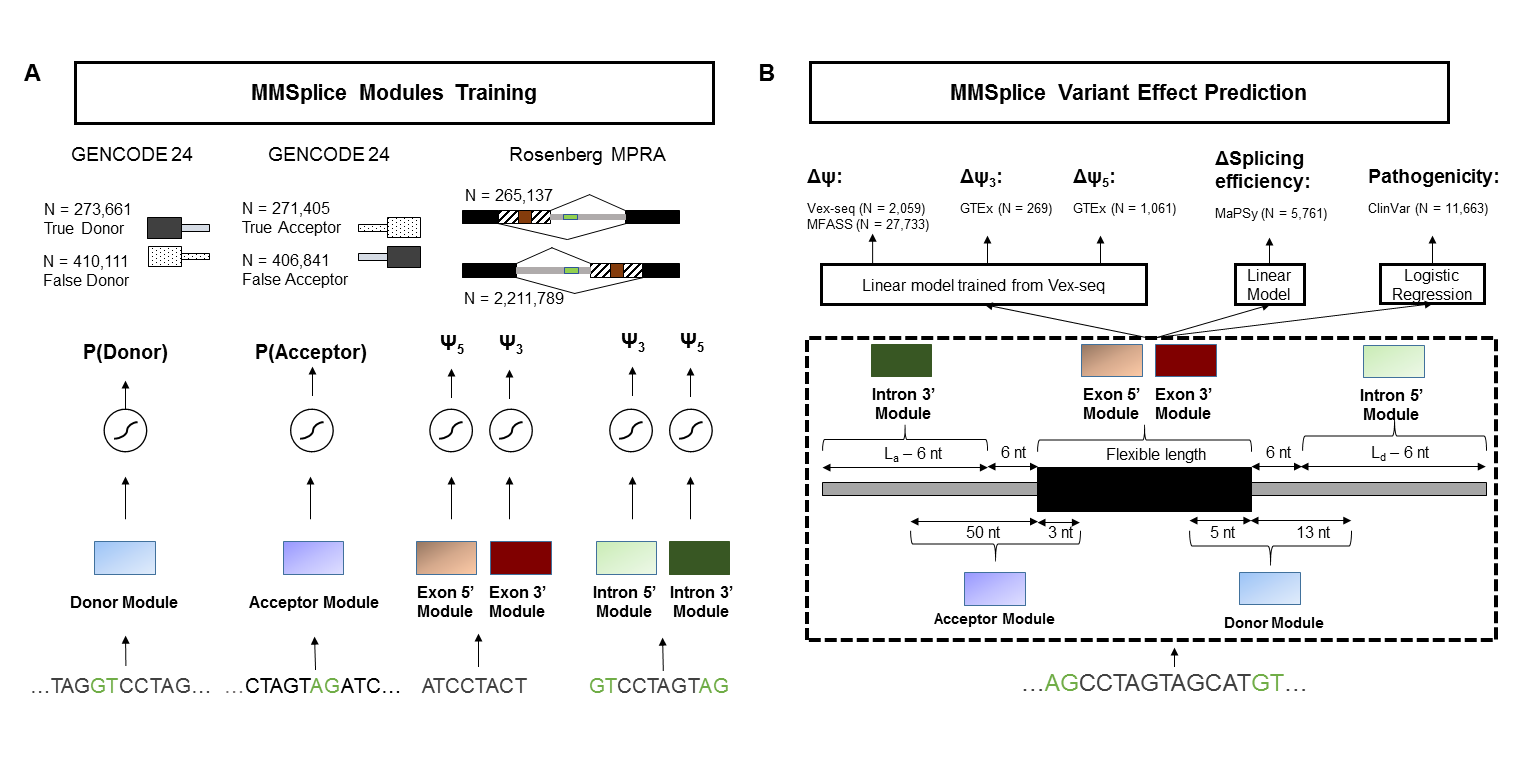
为了评分变异(包括插入和缺失),我们建议主要使用 deltaLogitPSI 预测,这是默认输出。差异剪接效率(dse)模型是从MMSplice模块和来自MaPSy的外显子变异训练的,因此只有外显子变异的预测进行了校准。
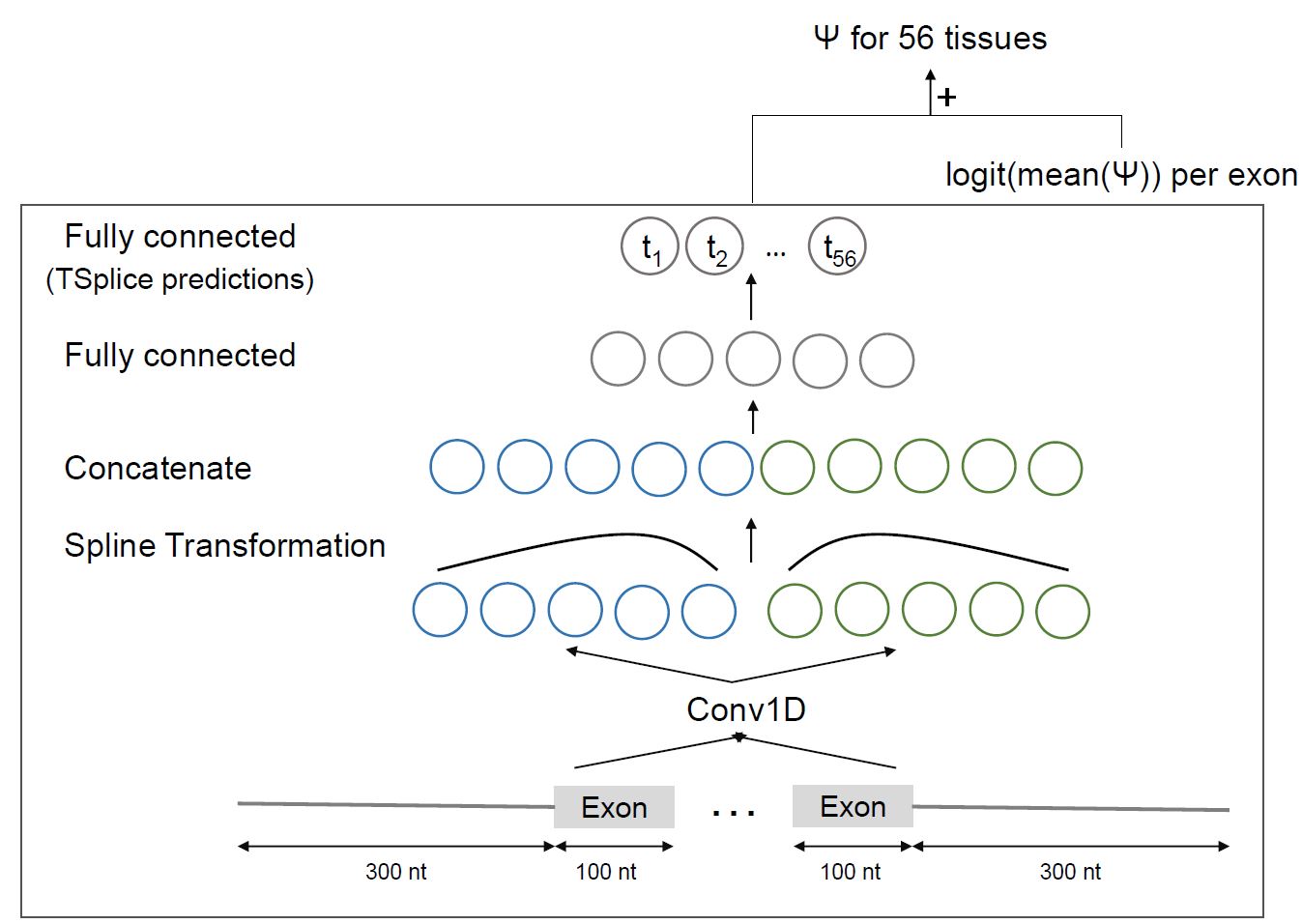
MTSplice 要使用MTSplice预测组织特异性变异效应,请在 SplicingVCFDataloader 中指定 tissue_specific=True。
# Import
from mmsplice.vcf_dataloader import SplicingVCFDataloader
from mmsplice import MMSplice, predict_save, predict_all_table
from mmsplice.utils import max_varEff
# example files
gtf = 'tests/data/test.gtf'
vcf = 'tests/data/test.vcf.gz'
fasta = 'tests/data/hg19.nochr.chr17.fa'
csv = 'pred.csv'
数据加载器用于从vcf加载变异
dl = SplicingVCFDataloader(gtf, fasta, vcf, tissue_specific=False)
为了预测组织特异性效应,请在数据加载器中使用 tissue_specific=True 代替
dl = SplicingVCFDataloader(gtf, fasta, vcf, tissue_specific=True)
使用默认MMSplice参数运行预测
# Specify model
model = MMSplice()
# Or predict and return as df
predictions = predict_all_table(model, dl, pathogenicity=True, splicing_efficiency=True)
要预测变异在 

dl = SplicingVCFDataloader(..., tissue_specific=True)
# Or predict and return as df
predictions = predict_all_table(model, dl, natural_scale=True)
一个变异可能映射到多个外显子。最后,我们将所有外显子的效应汇总为最大值。
# Summerize with maximum effect size
predictionsMax = max_varEff(predictions)
输出
MMSplice的输出是表格数据,包含以下描述的列
ID:变异的id字符串delta_logit_psi:MMSplice预测的主要得分,表示变异对外显子包含水平(PSI百分拼接入)的影响。得分在logit尺度上。如果得分是正的,则表示变异导致外显子更高的包含率。如果得分是负的,则表示变异导致外显子更高的排除率。如果delta_logit_psi大于2或小于-2,则可以认为变异的效应是显著的。exons:受变异影响的包含率受影响的外显子的遗传位置exon_id:受变异影响的包含率受影响的外显子的遗传idgene_id:外显子所属的基因的遗传idgene_name:外显子所属的基因的名称transcript_id:外显子所属的转录本的遗传idref_acceptorIntron:参考序列的接受体内含子得分ref_acceptor:参考序列的接受体得分ref_exon:参考序列的外显子得分ref_donor:参考序列的供体得分ref_donorIntron:参考序列的供体内含子得分alt_acceptorIntron:变异序列的接受体内含子得分alt_acceptor:变异序列的接受体得分alt_exon:变异序列的外显子得分alt_donor:变异序列的供体得分alt_donorIntron:变异序列的供体内含子得分pathogenicity:变异的潜在致病效应efficiency:变异对外显子剪接效率的影响
VEP插件
VEP插件包装了来自 mmsplice python包的预测函数。请查阅VEP插件的文档 在VEP_plugin/README.md下。
======= 历史
1.0.0 (2019-07-23)
- 依赖关系修复 #16
- 验证gtf、fasta、vcf染色体注释 #15
- 将预构建的外显子集与mmsplice一起分发。 #12
- 使用pyranges加快变异重叠。 #11
- 在外显子模块中更新掩码,进行批量预测
0.1.0 (2018-07-17)
- 首次在PyPI上发布。
项目详情
下载文件
下载适合您平台的文件。如果您不确定选择哪个,请了解更多关于安装包的信息。
源分发
构建分发
mmsplice-2.4.0.tar.gz的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | e467f5a96485afe1fbd01ab91dc22261df9c256fb67c4bc0d8fb38017b99c0ed |
|
| MD5 | d6cf4da2f6a3f9a93fa2c584455dedb8 |
|
| BLAKE2b-256 | 32bd3154f9e2979b2e7a35a2f3bcf071c12c9acced59b6d25415dcb9a9c6efc3 |
mmsplice-2.4.0-py2.py3-none-any.whl的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | d70a3a281b9433b89e6a8612cc2ca2611f54c35b90f46e16772b2868ec94b60a |
|
| MD5 | d2d23cbe10c30a54d8ed2446a9b1042e |
|
| BLAKE2b-256 | 14aa2bd61394b483d56b0d9011921e785d019848216336581b3144a21faff124 |