miniwdl AWS后端(Batch+EFS)
项目描述
miniwdl AWS插件
扩展 miniwdl 以在 AWS Batch 和 EFS 上运行工作流程
此miniwdl插件允许它将WDL任务作为AWS Batch作业执行。它使用EFS进行工作进度文件I/O,可选地将最终工作流程输出上传到S3。
在深入研究之前,请先考虑 AWS HealthOmics,它包括一个 用于WDL工作流程的托管服务,无需您配置所有基础设施。我们的配套项目 miniwdl-omics-run 提供了一个方便的CLI,用于使用本地WDL源代码文件启动HealthOmics运行。
有几种方法可以部署此miniwdl-aws插件
Amazon Genomics CLI
Amazon Genomics CLI 可以将所有必要的基础设施的 miniwdl-aws上下文 部署到您的AWS账户中。
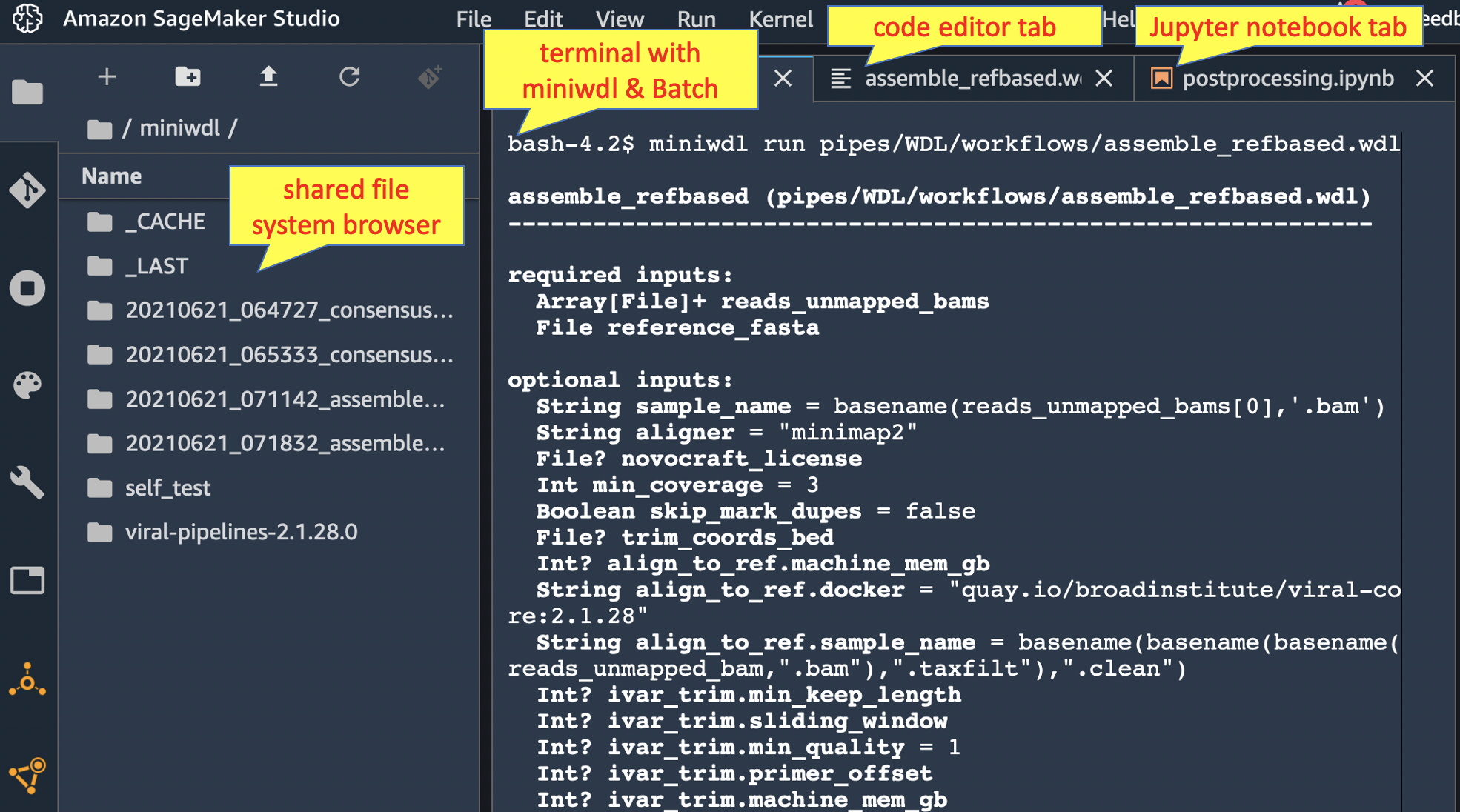
Amazon SageMaker Studio
或者,尝试使用 miniwdl-aws-studio 脚本来在 Amazon SageMaker Studio(一个具有终端和文件系统浏览器的Web IDE)中安装miniwdl进行交互式使用。您可以使用终端操作AWS Batch,使用文件系统浏览器管理EFS上的输入和输出,并使用Jupyter笔记本进一步分析输出。
使用自定义基础设施的 miniwdl-aws-submit
最后,高级操作员可以使用miniwdl-aws-terraform来部署/自定义必要的AWS基础设施,包括VPC、EFS文件系统、Batch队列和IAM角色。
在此方案中,本地命令行包装器miniwdl-aws-submit 在它自己的小型Batch作业中启动miniwdl来编排工作流程。然后,这个工作流程作业根据需要生成WDL 任务作业,而无需提交笔记本电脑在整个过程中保持连接。工作流程作业在轻量级的Fargate资源上运行,而任务作业在EC2 spot实例上运行。
提交工作流程作业
部署miniwdl-aws-terraform后,在本地执行pip3 install miniwdl-aws,使miniwdl-aws-submit程序可用。尝试自我测试
miniwdl-aws-submit --self-test --follow --workflow-queue miniwdl-workflow
然后启动一个病毒基因组组装,预计运行10-15分钟
miniwdl-aws-submit \
https://github.com/broadinstitute/viral-pipelines/raw/v2.1.28.0/pipes/WDL/workflows/assemble_refbased.wdl \
reads_unmapped_bams=https://github.com/broadinstitute/viral-pipelines/raw/v2.1.19.0/test/input/G5012.3.testreads.bam \
reference_fasta=https://github.com/broadinstitute/viral-pipelines/raw/v2.1.19.0/test/input/ebov-makona.fasta \
sample_name=G5012.3 \
--workflow-queue miniwdl-workflow \
--s3upload s3://MY-BUCKET/assemblies \
--verbose --follow
命令行类似于miniwdl run,但额外包含AWS相关参数
--workflow-queue要安排工作流程作业的Batch作业队列;miniwdl-aws-terraform的输出,默认为miniwdl-workflow。(也由环境变量MINIWDL__AWS__WORKFLOW_QUEUE设置)--follow实时流式传输工作流程日志而不是立即提交后退出。(--wait在无流式传输日志的情况下阻止工作流程。)--s3upload(可选)上传工作流程产品(包括日志和输出文件)的S3文件夹URI。必须在该miniwdl-aws-terraform部署中允许列表中。- 除非
--s3upload以/结尾,否则会在上传URI前缀中添加一个额外的子文件夹,等于miniwdl的自动时间戳前缀的运行名称。如果它以/结尾,则上传直接进入/下该文件夹(并且重复调用会预期覆盖它们)。
- 除非
miniwdl-aws-submit根据设置在工作流程队列上的标记检测其他基础设施细节(任务队列、EFS访问点、IAM角色);有关覆盖这些默认值的附加选项,请参阅miniwdl-aws-submit --help。
不被miniwdl-aws-submit消耗的参数传递到工作流程作业内部的miniwdl run;同样还有以MINIWDL__开头的环境变量,允许覆盖任何miniwdl配置选项(使用--no-env禁用)。有关在流程作业容器中预配置的选项,请参阅miniwdl_aws.cfg,其中一些可以调整以适应特定的负载。例如,要将miniwdl调用AWS Batch SubmitJob API的最大速率减半,请在miniwdl-aws-submit环境中设置MINIWDL__AWS__SUBMIT_PERIOD=2。
如果指定的WDL源代码是现有的本地.wdl或.zip文件,miniwdl-aws-submit会自动将其与工作流程作业一起作为要执行的WDL发送。对于.wdl文件,它运行miniwdl zip来检测并包含任何导入的WDL文件;而它假设.zip文件也是由miniwdl zip生成的。如果源代码太大,无法适应AWS Batch请求有效载荷(约50KB),则您需要通过引用将其传递到URL或EFS路径。
工作流程和任务作业都在/mnt/efs上挂载EFS。尽管通常使用HTTPS或S3 URI指定工作流程输入文件,但可以使用EFS上已存在的文件的/mnt/efs路径(这些路径可能不在提交机器上本地存在)。与WDL源代码不同,miniwdl-aws-submit不会尝试发送/上传本地输入文件。
EFS上的运行目录
Miniwdl在/mnt/efs/miniwdl_run下的目录中运行工作流程(使用--dir覆盖)。输出也保留在那里,以便在未来的运行中潜在地重用(为了避免,请使用--no-cache或擦除/mnt/efs/miniwdl_run/_CACHE)。
考虑到以EFS为中心的I/O模型,您需要一种远程浏览和管理文件系统内容的方法。配套的食谱lambdash-efs是一个选项;miniwdl-aws-terraform输出部署所需的配置详细信息(选择任何子网)。或者,设置一个挂载您的EFS的实例/容器,通过SSH或Web应用程序(例如JupyterHub、Cloud Commander、VS Code server)进行访问。
您还可以通过设置miniwdl-aws-submit --s3upload和
--delete-after success来自动清理EFS运行目录--delete-after failure在失败后删除目录--delete-after always在两种情况下都删除它- (或设置环境变量
MINIWDL__AWS__DELETE_AFTER_S3_UPLOAD)
在成功后删除运行目录可以防止输出在未来的运行中被重用。在失败后删除它可能会使调试更加困难(尽管日志被保留,见下文)。
关于文件系统隔离的安全提示
通过AWS Batch & EFS,miniwdl无法强制实施本地所实施的WDL任务容器之间的严格文件系统隔离。所有AWS Batch容器都有对整个EFS文件系统的读写访问权限(通过访问点查看),而不仅仅是它们的初始工作目录。
这通常是无害的,因为WDL任务应该只读取它们声明的输入并将其写入各自的相应工作/临时目录。但编写不良或恶意编写的任务可能会读取EFS上的其他文件并写入,甚至更改它们自己的输入文件或其他任务的输入文件。这可能导致不可预见的副作用或更严重的安全风险。
为了减轻这种情况,请使用本地后端彻底测试工作流程,该后端严格隔离任务容器的文件系统。如果WDL任务坚持就地修改输入文件,则可以使用--copy-input-files来解除它们(这将付出时间、空间和IOPS的代价)。最后,避免使用不受信任的WDL代码或容器映像;但如果它们是必要的,则使用单独的EFS访问点并适当地限制AWS Batch容器的IAM和网络配置。
EFS性能考虑因素
为了扩展到更大的工作负载,研究AWS关于EFS的性能和监控文档非常重要。像任何网络文件系统一样,EFS对吞吐量和IOPS的限制可能导致瓶颈,在最坏的情况下有效地冻结工作流程。
管理技巧
- 在AWS控制台的EFS区域监控文件系统吞吐量限制、IOPS和突发信用额(如果适用)。
- 保留默认的Elastic吞吐量模式(尽管它可能比其他模式更贵)
- 编写WDL任务以将任何纯临时文件写入
/tmp,这可能会使用本地缓存空间,而不是EFS工作目录。 - 配置miniwdl和AWS Batch以限制并发作业的数量和/或它们周转的速度(有关详细信息,请参阅miniwdl_aws.cfg)。
- 在时间上或跨越多个EFS文件系统将单独的工作流程运行分散开来。
FSx for Lustre和其他共享文件系统
如果EFS性能仍然不足,则可以将您的Batch计算环境配置为在实例启动时自动挂载其他共享文件系统。然后使用miniwdl-aws-submit --no-efs来使它假定文件系统将已挂载在某个位置(默认--mount /mnt/net)跨所有实例。在这种情况下,工作流程作业的计算环境预计将使用EC2而不是Fargate资源(通常需要挂载)。
miniwdl-aws-terraform 仓库包含一个变体设置,使用 FSx for Lustre 来配置。FSx 提供更高的吞吐量可伸缩性,但与 EFS 相比也有其他缺点(更高的初始成本、手动容量扩展、单个可用区部署、AWS 服务集成较少)。
日志和故障排除
如果终端日志不可用(通过 Studio 或 miniwdl-submit-awsbatch --follow)以跟踪工作流程失败,请在 EFS 或 S3 上运行的目录中查找 miniwdl 的常规日志文件。
每个任务作业的日志也会转发到 CloudWatch Logs 下的 /aws/batch/job 组和 miniwdl 日志中报告的日志流名称。使用 miniwdl-aws-submit,工作流程作业的日志也会转发。CloudWatch Logs 通过 AWS 控制台和 API 索引日志以进行结构化搜索。
配置错误的基础设施可能会阻止日志写入 EFS 或 CloudWatch。在这种情况下,使用 AWS Batch 控制台/API 查找工作流程或任务作业的状态消息。
任务可以通过设置 MINIWDL__LOG_TASK_USAGE__PERIOD=60(每 60 秒报告一次,或按需设置)在其标准错误日志中自行报告 CPU 和内存使用情况。使用 --verbose --follow 提交,或在任何任务的 CloudWatch Logs 流或 stderr.txt 文件中查看“容器使用”日志消息。
GPU 作业
Miniwdl-aws 识别任务 runtime{} 部分中的 gpu: true 设置,并将其转换为 AWS Batch 的 GPU 资源要求。要安排作业,批处理计算环境必须提供 GPU 实例类型。
默认情况下,gpu: true 转换为单个 GPU 的要求。WDL 规范定义了这是一个布尔值,因此无法明确请求给定任务的多个 GPU。可以将配置 MINIWDL__AWS__GPU_VALUE 设置为整数 N,以使所有具有 gpu: true 的任务都要求 N 个 GPU。
或者,miniwdl-aws 还识别 AWS HealthOmics 中使用的 acceleratorType 和 acceleratorCount 属性。任何以 "nvidia" 开头的 acceleratorType 都转换为 Batch GPU 要求;实际 GPU 类型将取决于计算环境提供的实例类型。
多 GPU 操作可能需要比 Batch 在每个任务容器中通常提供的更多共享内存。为了增加可用的共享内存,可以设置例如 MINIWDL__AWS__CONTAINER_PROPERTIES='{"linuxParameters":{"sharedMemorySize":4096}}'
贡献
欢迎拉取请求!如需帮助,在此打开一个问题或在 #miniwdl 在 OpenWDL Slack 中加入我们。
代码格式化和代码检查。 要准备代码以通过 CI 检查,
pip3 install --upgrade -r test/requirements.txt
pre-commit run --all-files
运行测试。 在 AWS 凭证终端会话中,
MINIWDL__AWS__WORKFLOW_QUEUE=miniwdl-workflow test/run_tests.sh
这将从当前代码修订版构建所需的 Docker 镜像并将其推送到 ECR 存储库(必须先用 aws ecr create-repository --repository-name miniwdl-aws 准备一次)。要测试来自 GitHub 公共注册表 或其他版本的镜像,请设置 MINIWDL__AWS__WORKFLOW_IMAGE 为所需的标签。
项目详情
miniwdl-aws-0.12.2.tar.gz 的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 8a130738c046a5f06382153278ab15e9bc0df00b0de4243036560d7a467ce52c |
|
| MD5 | 13879959c5519bd402b9172f610b36b8 |
|
| BLAKE2b-256 | a27451a0a9f223dbfd65439339a91d71a92366a6a2a3a5b68cf820427f1b5fd1 |