实验室动物单目3D姿态估计
项目描述
LiftPose3D

LiftPose3D是一个将2D姿态转换为实验室动物3D坐标的工具。基于三角测量的传统方法需要从多个相机同步获取,并需要复杂的校准协议。相比之下,LiftPose3D可以从单个相机的2D姿态重建3D姿态,在某些情况下,甚至不需要知道相机的位置或使用的镜头类型。有关理论背景和详细信息,请参阅我们的论文。
要训练LiftPose3D,理想情况下您需要(A)一个3D姿态库,(B)您将用于提升的相机的相应2D姿态,以及(C)相机矩阵(外参数和内参数)。
如果您无法访问
- (A)则使用提供的其中一个数据集,
- (B)则通过使用您的相机矩阵进行投影来获取2D图像(您需要校准以获得这些图像),
- (C)然后将您的相机放置得更远一些,假设弱透视。
启动
数据格式
在训练过程中,LiftPose3D接受形状为[N J 2]和[N J 3]的两个numpy数组作为输入和输出。在这里,N是姿态的数量,J是关节的数量。如果您有多个实验,您可以将数据作为字典提供,其中键是字符串,值是numpy数组。您还需要设置至少一个根关节和每个根关节的目标集。网络将预测目标集中的关节相对于根关节的位置。
对于每个示例,我们提供了一个独特的load.py文件,用于将数据转换为所需的[N J 3] LifPose3D格式。
训练
您可以使用以下通用语法通过实验1进行训练和实验2进行测试来训练网络。
from liftpose.main import train as lp3d_train
import numpy.random.rand
n_points, n_joints = 100, 5
train_2d, test_2d = rand((n_points, n_joints, 2)), rand((n_points, n_joints, 2))
train_3d, test_3d = rand((n_points, n_joints, 3)), rand((n_points, n_joints, 3))
train_2d = {"experiment_1": train_2d}
train_3d = {"experiment_1": train_3d}
test_2d = {"experiment_2": test_2d}
test_3d = {"experiment_2": test_3d}
roots = [0]
target_sets = [1,2,3,4]
lp3d_train(train_2d, test_2d, train_3d, test_3d, roots, target_sets)
默认情况下,输出将保存在相对于LiftPose3D调用路径的out文件夹中。您可以使用train函数的output_folder参数更改此行为。您可以在这里查看train函数的其他默认值和更长的文档。
例如,您可以通过在train函数中传递额外的参数training_kwargs来进一步配置训练。
from liftpose.main import train as lp3d_train
training_kwargs={ "epochs": 15, # train for 15 epochs
"resume": True, # resume training where it was stopped
"load" : 'ckpt_last.pth.tar'}, # use last training checkpoint to resume
lp3d_train(train_2d, test_2d, train_3d, test_3d, roots, target_sets, training_kwargs=training_kwargs)
您可以使用training_kwargs覆盖liftpose.lifter.opt内部的所有参数。
训练增强
增强训练数据是考虑数据集变异性的好方法,尤其是在训练数据稀缺时。
目前,在liftpose.lifter.augmentation中可用的选项有:
add_noise:在2D训练姿态上添加高斯噪声,以考虑测试时间的不确定性random_project:在测试时间相机方向未知的情况下,对3D姿态进行随机投影(训练将忽略输入train_2d)perturb_pose:当存在大动物到动物的变化时,通过改变段长度进行姿态增强project_to_cam:在测试时间相机矩阵不同且已知的情况下,对3D姿态进行确定性投影
可以在augmentation参数中指定训练增强选项,并且可以组合使用。
from liftpose.main import train as lp3d_train
from liftpose.lifter.augmentation import random_project
angle_aug = {'eangles' : {0: [[-10,10], [-10, 10], [-10,10]]}, #range of Euler angles (dictionary indexed by an integer which specifies the camera identify)
'axsorder': 'zyx', # order of rotations for euler angles
'vis' : None, # used in case not all joints are visible from a given camera
'tvec' : None, # camera translation vector
'intr' : None} # camera intrinsic matrix
aug = [random_project(**angle_aug)]
lp3d_train(train_2d, test_2d, train_3d, test_3d, roots, target_sets, aug=aug)
有关增强实现的示例,请参阅角度不变果蝇提升。
检查训练
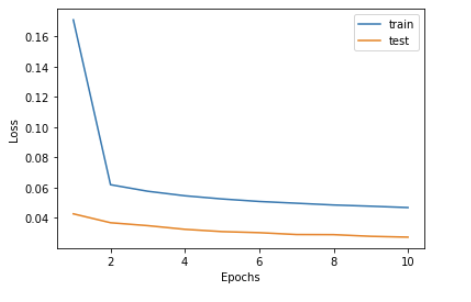
训练信息保存在train_log.txt中,可以如下可视化。
from liftpose.plot import read_log_train, plot_log_train
epoch, lr, loss_train, loss_test, err_test = read_log_train(par['out_dir'])
plot_log_train(plt.gca(), loss_train, loss_test, epoch)
这将绘制训练和测试损失。
测试网络
要测试在lp3d_train调用期间提供的数据,运行
from liftpose.main import test as lp3d_test
lp3d_test(par['out_dir'])
结果将保存在test_results.pth.tar文件中。
要测试新数据中的网络,运行
liftpose3d_test(par['out_dir'], test_2d, test_3d)
在这里,您提供上述描述的test_2d和test_3d。这将覆盖先前的test_results.pkl文件(如果有的话)。
我们还提供了一个简单的接口来从test_results.pkl文件加载测试结果。
from liftpose.postprocess import load_test_results
test_3d_gt, test_3d_pred, _ = load_test_results(par['out_dir'])
这将返回两个numpy数组:test_3d_gt,与test_3d相同,和test_3d_pred,其中包含LiftPose3D的预测。
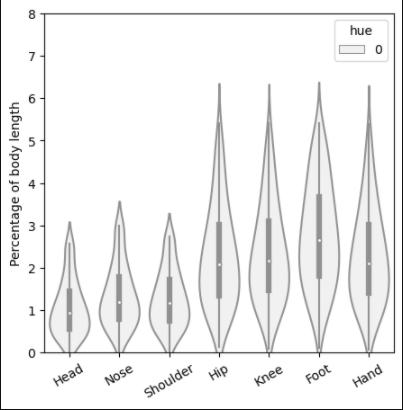
要生成误差分布,运行
from liftpose.plot import violin_plot
names = ['Head', 'Nose', 'Shoulder', 'Hip', 'Knee', 'Foot', 'Hand']
violin_plot(plt.gca(), test_3d_gt=test_3d_gt, test_3d_pred=test_3d_pred, test_keypoints=np.ones_like(test_3d_gt),
joints_name=names, units='m', body_length=2.21)
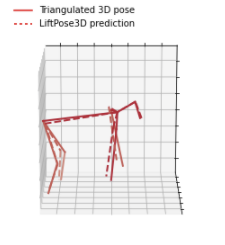
可视化3D姿态
要可视化输出3D姿态,首先在params.yaml文件中指定一个动物骨骼。请注意,骨骼信息或连接的关节仅用于可视化,而不是在训练中使用。您可以更详细地查看plot_pose_3d函数,了解如何在绘图期间使用骨骼和颜色参数。
data:
roots : [0]
target_sets : [[1, 2, 3, 4]]
vis:
colors : [[186, 30, 49]]
bones : [[0, 1], [1, 2], [2, 3], [3, 4]]
limb_id : [0, 0, 0, 0, 0]
我们提供了以下函数来可视化3D数据
from liftpose.plot import plot_pose_3d
fig = plt.figure(figsize=plt.figaspect(1), dpi=100)
ax = fig.add_subplot(111, projection='3d')
ax.view_init(elev=-75, azim=-90)
t = 0
plot_pose_3d(ax=ax, tar=test_3d_gt[t],
pred=test_3d_pred[t],
bones=par_data["vis"]["bones"],
limb_id=par_data["vis"]["limb_id"],
colors=par_data["vis"]["colors"],
legend=True)
这将输出类似的内容
您也可以轻松创建电影
from liftpose.plot import plot_video_3d
fig = plt.figure(figsize=plt.figaspect(1), dpi=100)
ax = fig.add_subplot(111, projection='3d')
ax.set_xlim([-4,2])
ax.set_ylim([-3,2])
ax.set_zlim([0,2])
plot_video_3d(fig, ax, n=gt.shape[0], par=par_data, tar=gt, pred=pred, trailing=10, trailing_keypts=[4,9,14,19,24,29], fps=20)
使用trailing来绘制具有所需长度的拖尾效果的点trailing_keypts。
使用点的子集进行训练
如果您想防止某些2D/3D点在训练中使用,可以将train_keypts参数传递到train函数中,该参数与train_3d具有相同的形状,但具有布尔数据类型。或者,如果您有缺失的关键点,可以将它们转换为np.NaN。在这两种情况下,这些点的损失将不会在反向传播中使用。
项目详情
下载文件
下载适用于您的平台的文件。如果您不确定选择哪个,请了解更多关于安装包的信息。
源代码发行版
构建发行版
liftpose-0.22.tar.gz 的散列
| 算法 | 散列摘要 | |
|---|---|---|
| SHA256 | 9994756752d983386850fb99dab000ddf4c3bbbed1806288cd6c83495f02ef55 |
|
| MD5 | 392235721bf90bdbd09fe9493853deab |
|
| BLAKE2b-256 | ed56d8538dd68175fa58d413e1ebe6c69af9982ba12027d436aee2f4d98e202a |
liftpose-0.22-py3-none-any.whl 的散列
| 算法 | 散列摘要 | |
|---|---|---|
| SHA256 | 4b78cdae95b7d1f6c1b3682dafcd979319ebb411a3f2dae89faf878610e6b4ba |
|
| MD5 | 2b46fbd13d209a1766c4e58aff7e763f |
|
| BLAKE2b-256 | a6b9491b5c593b5ddcf42d1be17add7156824c45dfc0d428876ec5ba9a6219f5 |



