Python包,用于懒加载生成用于Keras模型训练的合成基因组序列。
项目描述
Python包,用于懒加载生成用于Keras模型训练的合成基因组序列。
如何安装此包?
像往常一样,只需使用pip下载即可
pip install keras_synthetic_genome_sequence测试覆盖率
由于一些处理覆盖率的软件有时会得到略有不同的结果,这里列出了三个结果
使用示例
要使用GapSequence来训练您的Keras模型,您需要获得您打算在合成间隙中模拟的生物学间隙的统计指标。
为此,此包提供了一个名为get_gaps_statistics的实用工具,允许您获取给定基因组组装中间隙的均值和协方差。
基因组组装自动从UCSC下载,使用ucsc_genomes_downloader,然后从中提取包含的间隙,并将它们的窗口扩展到给定的一个,在过滤给定max_gap_size之后,因为您可能希望将间隙大小限制在一个相对较小的一个(例如,间隙可以长到数万个核苷酸,如在端粒中)。
让我们先列出所有重要的参数
assembly = "hg19"
window_size = 200
batch_size = 128现在我们可以开始检索间隙统计信息
from keras_synthetic_genome_sequence.utils import get_gaps_statistics
number, mean, covariance = get_gaps_statistics(
assembly=assembly,
max_gap_size=100,
window_size=window_size
)
print("I have identified {number} gaps!".format(number=number))现在您必须选择要应用合成间隙的基准,例如基因组组装hg19中无间隙的区域,染色体chr1。这些区域将必须被压缩成与您选择的间隙统计窗口_size形状兼容的小块。我们可以按以下方式检索这些区域
from ucsc_genomes_downloader import Genome
from ucsc_genomes_downloader.utils import tessellate_bed
genome = Genome(assembly, chromosomes=["chr1"])
ground_truth = tessellate_bed(genome.filled(), window_size=window_size)获得的数据帧将具有床状格式,如下所示
chrom |
chromStart |
chromEnd |
|
|---|---|---|---|
0 |
chr1 |
10000 |
10200 |
1 |
chr1 |
10200 |
10400 |
2 |
chr1 |
10400 |
10600 |
3 |
chr1 |
10600 |
10800 |
4 |
chr1 |
10800 |
11000 |
现在我们准备好实际创建GapSequence了
from keras_synthetic_genome_sequence import GapSequence
gap_sequence = GapSequence(
assembly=assembly,
bed=ground_truth,
gaps_mean=mean,
gaps_covariance=covariance,
batch_size=batch_size
)现在,拥有一个输入输出形状为(batch_size,window_size,4)的模型,我们可以按以下方式训练它
model = build_my_denoiser()
model.fit_generator(
gap_sequence,
steps_per_epoch=gap_sequence.steps_per_epoch,
epochs=2,
shuffle=True
)快乐去噪!
生物与合成分布的比较
以下图像表示hg19、hg38、mm9和mm10基因组组装中gap的生物和合成分布,考虑长度上限为100核苷酸,总窗口大小为1000。用于将多元高斯分布转换为整数的阈值是0.4,这是python包中的默认值。
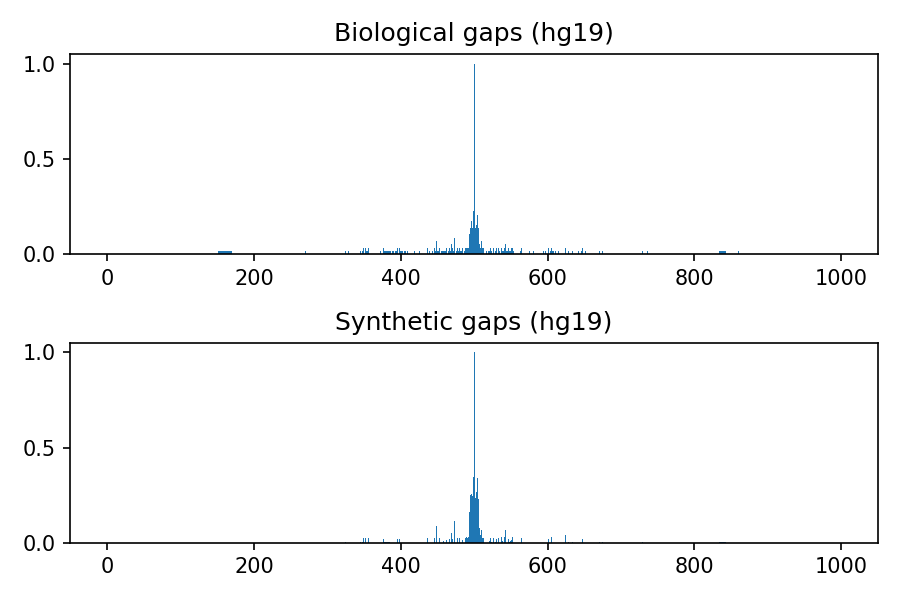
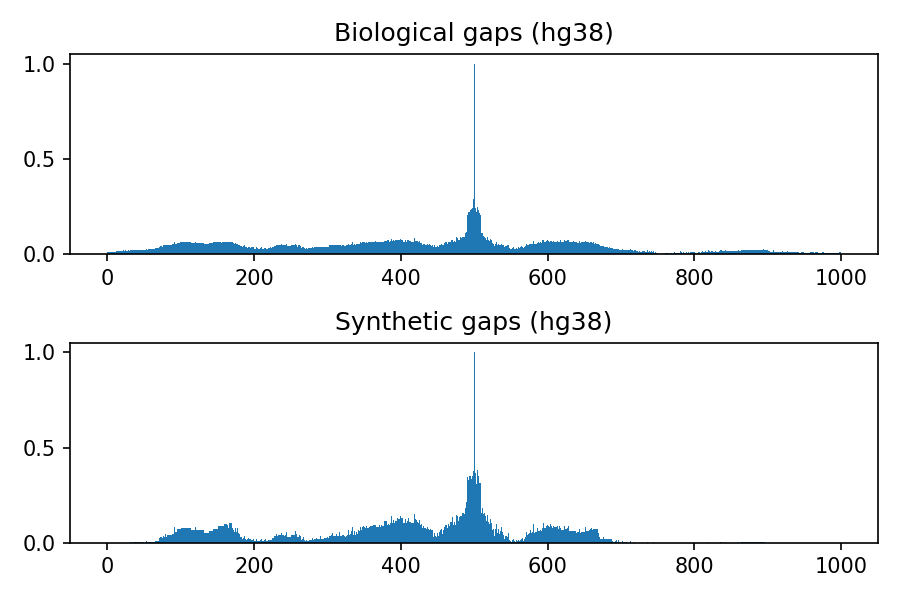
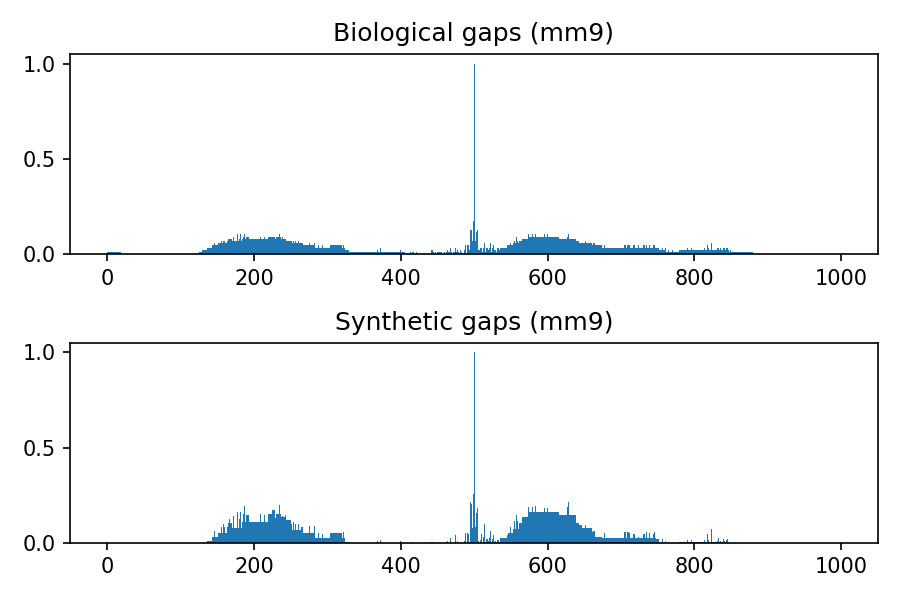
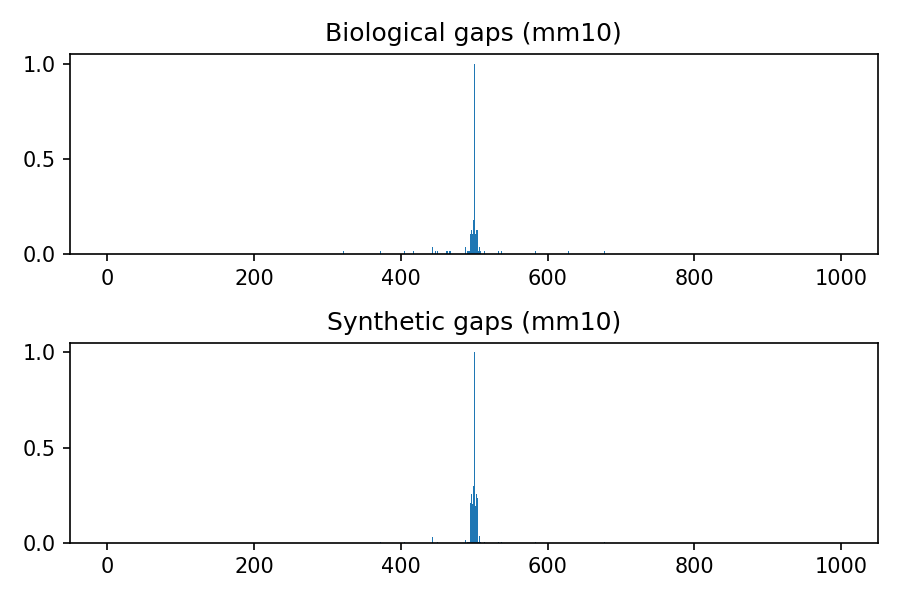
项目详情
keras_synthetic_genome_sequence-1.1.4.tar.gz的散列
| 算法 | 散列摘要 | |
|---|---|---|
| SHA256 | b54d05c0cd1818357d6ebfdcf65aba13a5bf9ac360a3170654e6fc47771357bf |
|
| MD5 | 1c8577d5ede1482217b821ce74cf9fcf |
|
| BLAKE2b-256 | 3affee98e41f735d95926c55fdc9b50f21fa406321e3773eb295d1a283b71403 |













