数字病理图像处理的Python库
项目描述

| 测试状态 |


|
| 代码质量 |




|
| 版本信息 |




|
| 许可证 |

|
| 文档 | 
|
兼容性细节
| 操作系统 | Python版本 |
|---|---|
| Linux |  |
| MacOs |  |
目录
动机
组织切片的病理学分析是评估许多复杂疾病(如肿瘤)存在并了解其本质的金标准。在日常工作实践中,病理学家通常对有限的区域进行组织切片显微镜检查,临床评估依赖于核形态、细胞分布和颜色(染色)等多个因素:这个过程耗时,可能导致信息丢失,且存在观察者之间的差异。
数字病理学的出现正在改变病理学家的工作方式和协作方式,并为计算病理学的新时代开辟了道路。特别是,组织病理学预计将成为医学AI革命的中心[1],这一预测得到了深度学习应用在数字病理学中不断取得成功的支持。
全切片图像(WSI)即组织切片从玻璃到数字格式的转换,从医疗和计算角度都是信息丰富的来源。WSI可以使用不同的染色技术(例如H&E或IHC)染色,并且通常非常大(每张切片高达几个GB)。由于WSI典型的金字塔结构,可以以不同的放大倍数检索图像,这提供了比颜色更深层次的信息。
然而,处理WSI远非简单。首先,WSI可以存储在不同的专有格式中,这取决于用于数字化切片的扫描仪,而且尚无标准协议。WSI也可能出现一些不必要的人工制品,如阴影、霉菌或注释(笔迹)。此外,鉴于其尺寸,不可能一次性处理整个WSI,例如,无法直接输入神经网络:需要裁剪较小的组织区域(瓦片),而这又需要组织检测步骤。
本项目的目的是提供一个工具,在可重复的环境中处理WSI,以支持临床和科学研究。histolab旨在处理WSI,自动检测组织,并检索信息丰富的瓦片,因此它可以集成到深度学习管道中。
入门指南
先决条件
请参阅安装说明。
文档
在此处阅读完整文档https://histolab.readthedocs.io/en/latest/。
交流
加入我们的用户组Slack。
5分钟简介
快速入门
在此,我们提供了一个使用histolab从示例WSI中提取瓦片数据集的逐步教程。相应的Jupyter Notebook可在https://github.com/histolab/histolab-box找到:此存储库包含一个完整的histolab环境,可以通过Docker在所有平台上使用。
因此,用户可以选择通过histolab-box或在其Python虚拟环境中安装histolab来使用它。在后一种情况下,由于histolab软件包已在PyPi上发布,因此可以通过以下命令轻松安装
pip install histolab
或者,可以通过conda安装
conda install -c conda-forge histolab
TCGA数据
首先,让我们导入一些数据以供工作,例如data模块中可用的前列腺组织切片和卵巢组织切片
from histolab.data import prostate_tissue, ovarian_tissue
注意:要使用data模块,您需要安装pooch,它也在PyPI上提供(https://pypi.ac.cn/project/pooch/)。如果我们在使用Vagrant/Docker虚拟环境,此步骤是不必要的。
调用data函数将自动从相应的存储库下载WSI并将其保存到缓存目录中
prostate_svs, prostate_path = prostate_tissue()
ovarian_svs, ovarian_path = ovarian_tissue()
注意,每个data函数输出相应的幻灯片,作为一个OpenSlide对象,以及保存幻灯片的路径。
幻灯片初始化
histolab将WSI文件映射到一个Slide对象。每次使用WSI都需要与slide模块中包含的Slide对象进行一对一的关联。
from histolab.slide import Slide
要初始化一个幻灯片,需要指定WSI路径,以及保存瓦片的processed_path。在我们的示例中,我们希望每个幻灯片的processed_path是当前工作目录的子文件夹。
import os
BASE_PATH = os.getcwd()
PROCESS_PATH_PROSTATE = os.path.join(BASE_PATH, 'prostate', 'processed')
PROCESS_PATH_OVARIAN = os.path.join(BASE_PATH, 'ovarian', 'processed')
prostate_slide = Slide(prostate_path, processed_path=PROCESS_PATH_PROSTATE)
ovarian_slide = Slide(ovarian_path, processed_path=PROCESS_PATH_OVARIAN)
注意:如果幻灯片存储在同一个文件夹中,可以使用slide模块的SlideSet对象直接在整个数据集上执行此操作。
使用Slide对象,我们可以轻松地检索有关幻灯片的信息,例如幻灯片名称、可用级别的数量、原生放大倍数或指定级别的尺寸。
print(f"Slide name: {prostate_slide.name}")
print(f"Levels: {prostate_slide.levels}")
print(f"Dimensions at level 0: {prostate_slide.dimensions}")
print(f"Dimensions at level 1: {prostate_slide.level_dimensions(level=1)}")
print(f"Dimensions at level 2: {prostate_slide.level_dimensions(level=2)}")
Slide name: 6b725022-f1d5-4672-8c6c-de8140345210
Levels: [0, 1, 2]
Dimensions at level 0: (16000, 15316)
Dimensions at level 1: (4000, 3829)
Dimensions at level 2: (2000, 1914)
print(f"Slide name: {ovarian_slide.name}")
print(f"Levels: {ovarian_slide.levels}")
print(f"Dimensions at level 0: {ovarian_slide.dimensions}")
print(f"Dimensions at level 1: {ovarian_slide.level_dimensions(level=1)}")
print(f"Dimensions at level 2: {ovarian_slide.level_dimensions(level=2)}")
Slide name: b777ec99-2811-4aa4-9568-13f68e380c86
Levels: [0, 1, 2]
Dimensions at level 0: (30001, 33987)
Dimensions at level 1: (7500, 8496)
Dimensions at level 2: (1875, 2124)
注意:如果幻灯片属性中提供了原生放大倍数(即扫描幻灯片时使用的放大倍数),还可以使用level_magnification_factor属性将所需的级别转换为相应的放大倍数。
print(
"Native magnification factor:",
prostate_slide.level_magnification_factor()
)
print(
"Magnification factor corresponding to level 1:",
prostate_slide.level_magnification_factor(level=1),
)
Native magnification factor: 20X
Magnification factor corresponding to level 1: 5.0X
此外,我们还可以在单独的窗口中检索或显示幻灯片缩略图。
prostate_slide.thumbnail
prostate_slide.show()
ovarian_slide.thumbnail
ovarian_slide.show()
瓦片提取
一旦定义了Slide对象,我们就可以进行提取瓦片。为了加快提取过程,histolab自动检测具有最大连接区域的组织区域,并在此区域内裁剪瓦片。tiler模块实现了不同的瓦片提取策略,并提供了一个直观的接口,以轻松检索适合我们任务的瓦片数据集。特别是,每种提取方法都可以使用几个常用参数进行自定义。
tile_size:瓦片大小;level:提取级别(从0到可用级别的数量);check_tissue:是否需要保存瓦片时至少有最小百分比的组织;tissue_percent:介于0.0和100.0之间的数字,表示图像总面积上所需的最小组织百分比(默认为80.0);prefix:要在瓦片文件名开头添加的前缀(默认为空字符串);suffix:要添加到瓦片文件名末尾的后缀(默认为.png)。
随机提取
我们可能采用的最简单的方法是从我们的幻灯片中随机裁剪固定数量的瓦片;在这种情况下,我们需要使用RandomTiler提取器。
from histolab.tiler import RandomTiler
假设我们想要从我们的前列腺幻灯片在级别2上随机提取30个128大小的正方形瓦片,并且我们只想保存至少有80%组织在内的瓦片。然后,我们按照以下方式初始化我们的RandomTiler提取器:
random_tiles_extractor = RandomTiler(
tile_size=(128, 128),
n_tiles=30,
level=2,
seed=42,
check_tissue=True, # default
tissue_percent=80.0, # default
prefix="random/", # save tiles in the "random" subdirectory of slide's processed_path
suffix=".png" # default
)
注意,我们还指定了随机种子,以确保提取过程的可重复性。
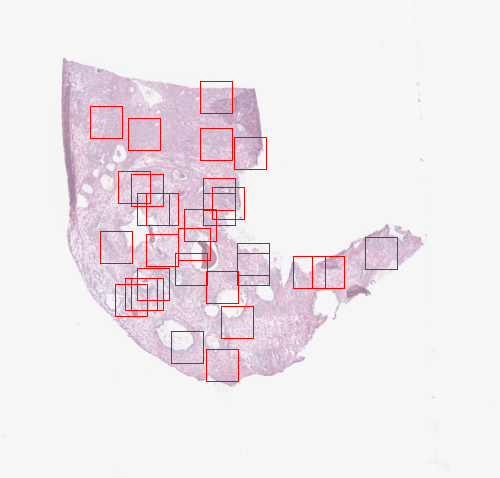
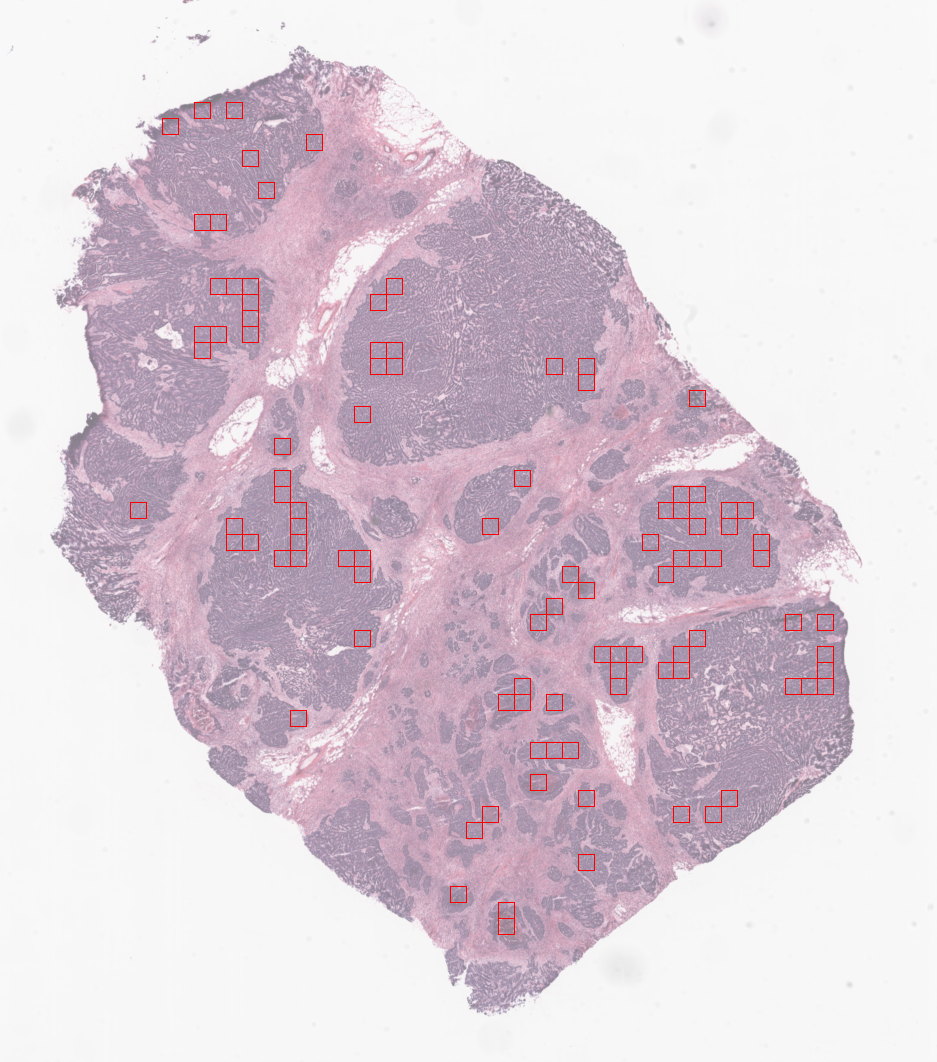
在开始提取过程并保存之前,我们可能想要检查tiler所选的瓦片;RandomTiler的locate_tiles方法返回一个带有相应瓦片轮廓的缩放幻灯片。还可以指定背景幻灯片的透明度和每个瓦片边框使用的颜色。
random_tiles_extractor.locate_tiles(
slide=prostate_slide,
scale_factor=24, # default
alpha=128, # default
outline="red", # default
)
启动提取就像在提取器上调用extract方法一样简单,并将幻灯片作为参数传递。
random_tiles_extractor.extract(prostate_slide)
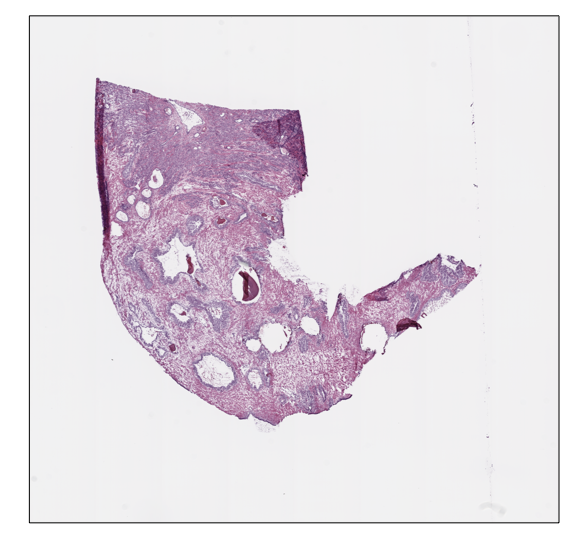
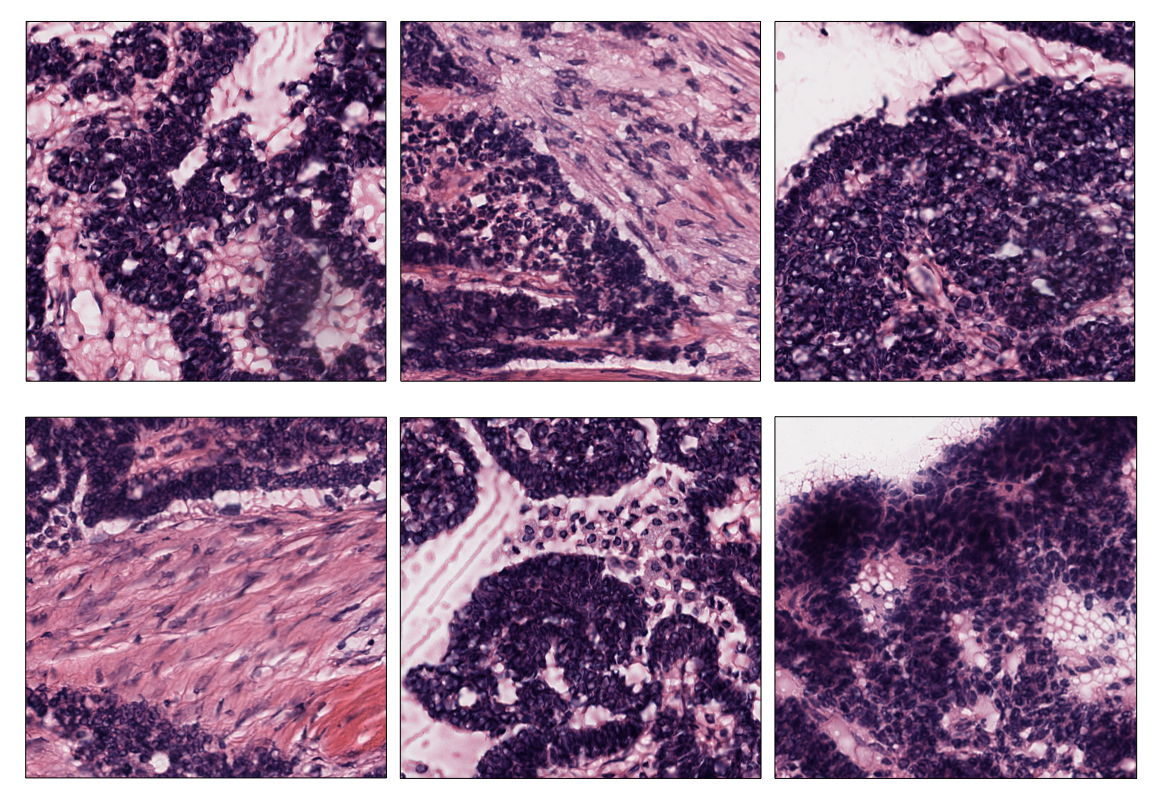
从前列腺幻灯片在级别2上提取的随机瓦片。
网格提取
除了随机选择瓦片,我们可能希望检索所有可用的瓦片。Grid Tiler提取器在WSI中检测到的最大组织区域上按照网格结构裁剪瓦片。
from histolab.tiler import GridTiler
在我们的示例中,我们希望从卵巢切片中提取大小为512的平方块,位于0级,独立于检测到的组织量。默认情况下,瓦片不会重叠,即定义两个相邻瓦片之间重叠像素数的参数pixel_overlap被设置为0。
grid_tiles_extractor = GridTiler(
tile_size=(512, 512),
level=0,
check_tissue=False,
pixel_overlap=0, # default
prefix="grid/", # save tiles in the "grid" subdirectory of slide's processed_path
suffix=".png" # default
)
同样,我们可以利用locate_tiles方法在切片的缩放版本上可视化选定的瓦片。
grid_tiles_extractor.locate_tiles(
slide=ovarian_slide,
scale_factor=64,
alpha=64,
outline="#046C4C",
)
grid_tiles_extractor.extract(ovarian_slide)
当在提取器上调用extract方法时,提取过程开始。
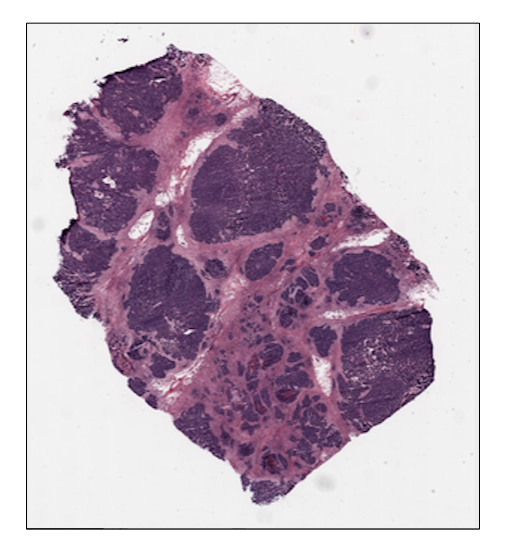
从卵巢切片0级提取的非重叠网格瓦片的示例。
基于分数的提取
根据我们将要使用的瓦片数据集的任务,提取的瓦片可能不会提供同等的信息。`ScoreTiler`允许我们仅保存所有具有网格结构的提取瓦片中的“最佳”瓦片,基于特定的评分函数。例如,假设我们的目标是检测卵巢切片上的有丝分裂活性。在这种情况下,具有较高核存在率的瓦片比核很少或没有的瓦片更受欢迎。我们可以利用`scorer`模块中的`NucleiScorer`函数按组织量和苏木精染色量对提取的瓦片进行排序。特别是,评分的计算公式为


首先,我们需要提取器和评分器。
from histolab.tiler import ScoreTiler
from histolab.scorer import NucleiScorer
由于`ScoreTiler`扩展了`GridTiler`提取器,我们还设置了`pixel_overlap`作为附加参数。此外,我们可以通过`n_tile`参数指定我们想要保存的前几名瓦片数量。
scored_tiles_extractor = ScoreTiler(
scorer = NucleiScorer(),
tile_size=(512, 512),
n_tiles=100,
level=0,
check_tissue=True,
tissue_percent=80.0,
pixel_overlap=0, # default
prefix="scored/", # save tiles in the "scored" subdirectory of slide's processed_path
suffix=".png" # default
)
请注意,`ScoreTiler`也实现了`locate_tiles`方法,该方法在切片的缩放版本上可视化具有最高评分的前n_tiles。
grid_tiles_extractor.locate_tiles(slide=ovarian_slide)
最后,当我们提取裁剪图像时,我们还可以将保存的瓦片及其评分写入CSV文件。
summary_filename = "summary_ovarian_tiles.csv"
SUMMARY_PATH = os.path.join(ovarian_slide.processed_path, summary_filename)
scored_tiles_extractor.extract(ovarian_slide, report_path=SUMMARY_PATH)
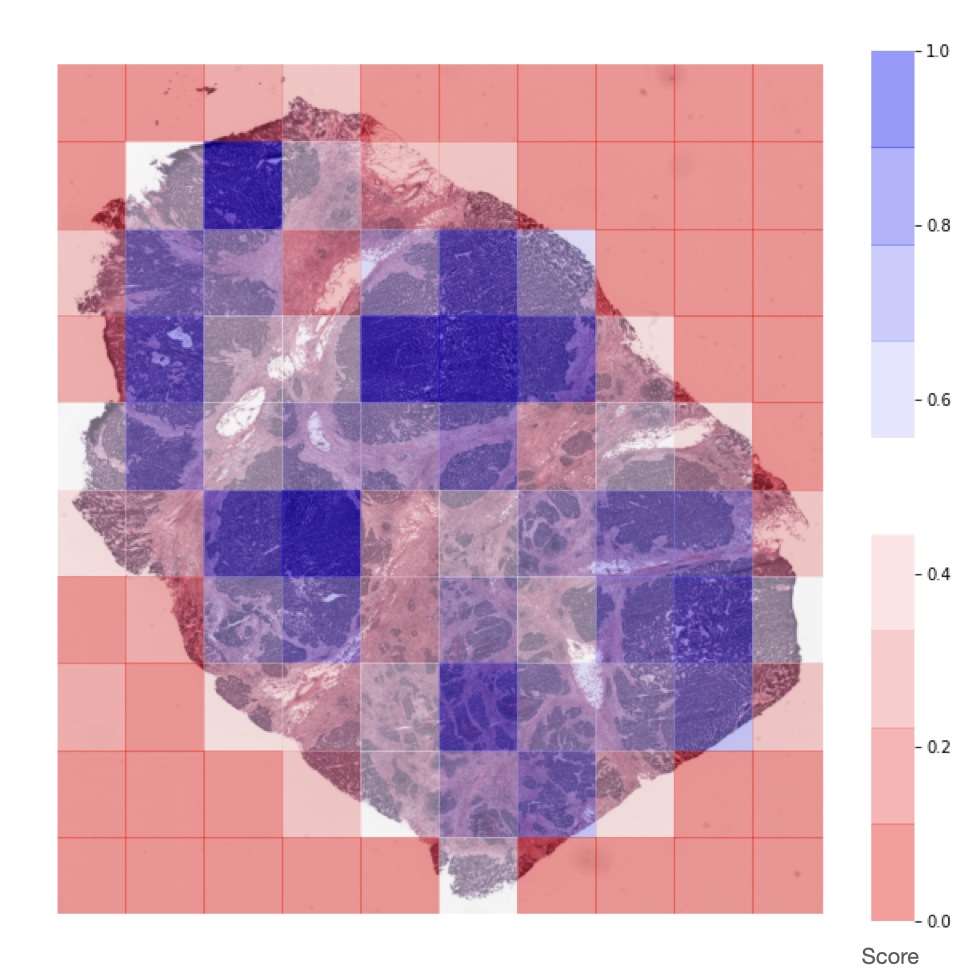
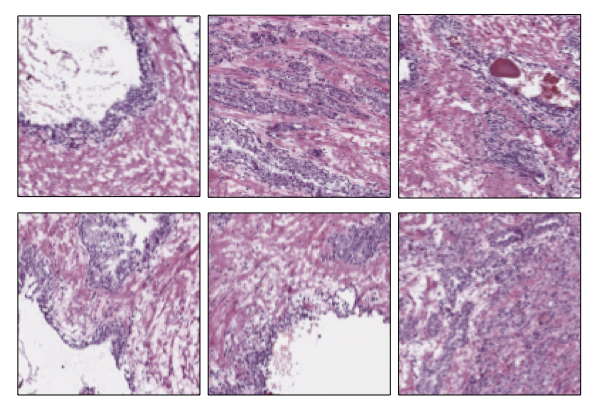
基于检测到的核数量,由`NucleiScorer`为每个提取的瓦片分配的评分表示。
版本管理
我们使用PEP 440进行版本控制。
作者
许可证
本项目受Apache License Version 2.0许可 - 有关详细信息,请参阅LICENSE.txt文件。
路线图
致谢
参考文献
[1] Colling, Richard, et al. "Artificial intelligence in digital pathology: A roadmap to routine use in clinical practice." The Journal of pathology 249.2 (2019)
贡献指南
如果您想为histolab做出贡献,请务必查看贡献指南。
项目详情
下载文件
下载适用于您平台的文件。如果您不确定选择哪个,请了解有关安装包的更多信息。