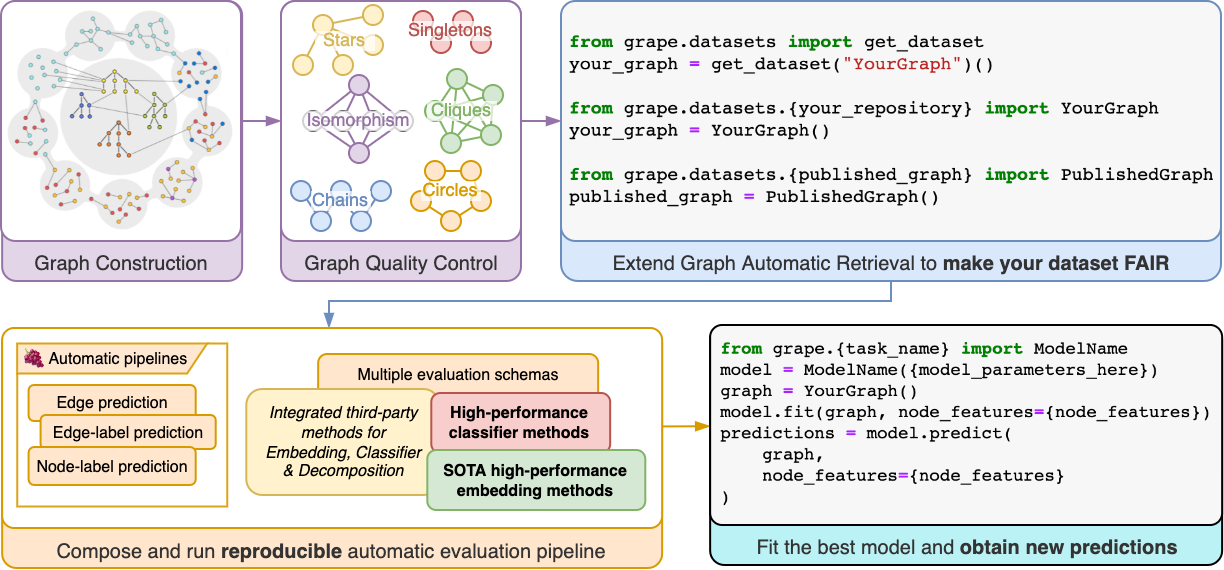
🍇 GRAPE 是一个用于预测和评估的 Rust/Python 图表示学习库。
项目描述
GRAPE (Graph Representation leArning, Predictions and Evaluation) 是一个快速图处理和嵌入库,旨在扩展到大型图,并可在台式机和笔记本电脑以及高性能计算工作站集群上运行。
该库是用 Rust 和 Python 编程语言编写的,由 AnacletoLAB (米兰大学计算机科学系) 开发,并与 Robinson 实验室 - 杰克逊基因组医学实验室 以及 BBOP - 伯克利国家实验室 合作开发。
该库由两个主要模块组成,Ensmallen,这是一个 Rust/Python 高性能图处理子模块,以及 Embiggen,这是一个 Python 图表示学习、预测和评估子模块。
GRAPE 的安装
通常,只需运行以下命令从 PyPi 安装它
pip install grape通常,安装不会超过一分钟。
可以在任何支持Rust和LLVM的操作系统、libc版本和CPU架构(例如Arm、AArch64、RiscV、Mips)上手动编译Ensmallen。如果您需要帮助,请打开一个问题。
库的主要功能
- 60+ 节点嵌入模型,易于集成第三方库。
DeepWalk、Walklets和基于Node2Vec的CBOW、SkipGram和GloVe,还包括度规范化!
非结构化、结构化嵌入、TransE
所有嵌入模型都有MMAPP版本,允许非常大的嵌入
所有嵌入模型都支持混合精度,允许使用16位浮点数进行嵌入
集成来自Karate Club和PyKEEN的模型
- 20+ 分类器模型,易于集成第三方库。
所有sklearn模型,适用于边缘预测、边缘标签预测和节点标签预测
GraphSAGE和Kipf GCN用于边缘预测、边缘标签预测和节点标签预测
- 图可视化工具
嵌入的TSNE、UMAP、PCA
边缘属性
教程
您可以在这里找到覆盖GRAPE库各个方面的教程。所有教程尽可能独立,可以在COLAB上立即执行。
如果您认为任何示例可能有所帮助,请随时在GitHub上打开一个问题,描述本教程中缺少的内容。
文档
在线文档(目前正在更新中)
库的在线文档可以在这里找到。由于Ensmallen是用Rust编写的,而PyO3(我们用于Python绑定的包)不支持类型,文档是通过生成一个空骨架包来获得的。这允许您拥有适当的文档,但您无法在其中看到源代码。
使用自动方法建议实用工具
为了帮助您使用该库,GRAPE提供了一个集成推荐系统,旨在帮助您找到一种方法,或者如果由于任何原因方法被重命名,找到其新名称。
例如,在加载了STRING 人源图之后,可以通过简单地输入components来检索计算连通组件的函数
from grape.datasets.string import HomoSapiens
graph = HomoSapiens()
graph.components上面的代码将引发以下错误,并建议具有相似或相关名称的方法
AttributeError Traceback (most recent call last)
<ipython-input-3-52fac30ac7f6> in <module>()
----> 2 graph.components
AttributeError: The method 'components' does not exists, did you mean one of the following?
* 'remove_components'
* 'connected_components'
* 'strongly_connected_components'
* 'get_connected_components_number'
* 'get_total_edge_weights'
* 'get_mininum_edge_weight'
* 'get_maximum_edge_weight'
* 'get_unchecked_maximum_node_degree'
* 'get_unchecked_minimum_node_degree'
* 'get_weighted_maximum_node_degree'在我们的例子中,用于计算图组件的方法将是connected_components。
现在获取方法文档的最简单方法是使用Python的帮助,如下所示
help(graph.connected_components)上述操作将返回以下内容
connected_components(verbose) method of builtins.Graph instance
Compute the connected components building in parallel a spanning tree using [bader's algorithm](https://www.sciencedirect.com/science/article/abs/pii/S0743731505000882).
**This works only for undirected graphs.**
The returned quadruple contains:
- Vector of the connected component for each node.
- Number of connected components.
- Minimum connected component size.
- Maximum connected component size.
Parameters
----------
verbose: Optional[bool]
Whether to show a loading bar or not.
Raises
-------
ValueError
If the given graph is directed.
ValueError
If the system configuration does not allow for the creation of the thread pool.引用GRAPE
如果该文档对您的研究有所帮助,请引用以下论文
@article{cappelletti2023grape,
title={GRAPE for Fast and Scalable Graph Processing and random walk-based Embedding},
author={Cappelletti, L. and Fontana, T. and Casiraghi, E. and Ravanmehr, V. and Callahan, T.J. and Cano, C. and Joachimiak, M.P. and Mungall, C.J. and Robinson, P.N. and Reese, J. and Valentini, G.},
year={2023},
doi={10.1038/s43588-023-00465-8},
journal={Nature Computational Science}}项目详情
grape-0.2.4.tar.gz的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 119ef66ea8486fae52a3e0a1b6658758e0723704805e9b069fccdfdeeab3692e |
|
| MD5 | 48004bc9f76e8c9c644a30a56db60c0c |
|
| BLAKE2b-256 | 550e9ca3f9a192fb950c5fd7ef6e8c87bc44d64d8f499da3f195d889ea972106 |