未提供项目描述
项目描述
GeneScape: 基因功能可视化
GeneScape 是一款用于可视化基因功能的软件工具。用户输入基因列表,软件随后绘制与基因相关的基因本体(GO)术语的子图。
GeneScape 是一个基于Python的 Shiny 应用程序,可以在命令行和图形用户界面中运行。
出版物
软件的Shiny版本可以通过以下网址访问
公共接口可能存在使用限制。要无限使用,请本地安装并运行软件。
本地安装
用户还可以通过使用 pip 安装软件在他们的系统上运行程序
pip install genescape
安装后,可以通过以下方式启动Shiny界面
genescape web
在浏览器中访问 https://:8000 URL 以查看界面。
命令行使用
程序也可以在命令行中使用以生成图像或注释
genescape tree绘制信息丰富的基因本体(GO)子图genescape annotate用GO功能注释基因列表genescape web为tree命令提供网络界面
GeneScape做什么?
GeneScape 的工作方式如下
- 首先从输入列表读取基因。
- 然后提取与输入基因相关的注释。
- 最后,基于这些注释构建并可视化功能子树。
注意:即使是很短的基因列表(不到十个基因)也可以创建大型树。可以通过最小覆盖度(有多少基因共享功能)或功能模式(匹配特定模式的功能)进行过滤。
当没有明确指定阈值时,GeneScape将尝试找到一个合理的覆盖度阈值。
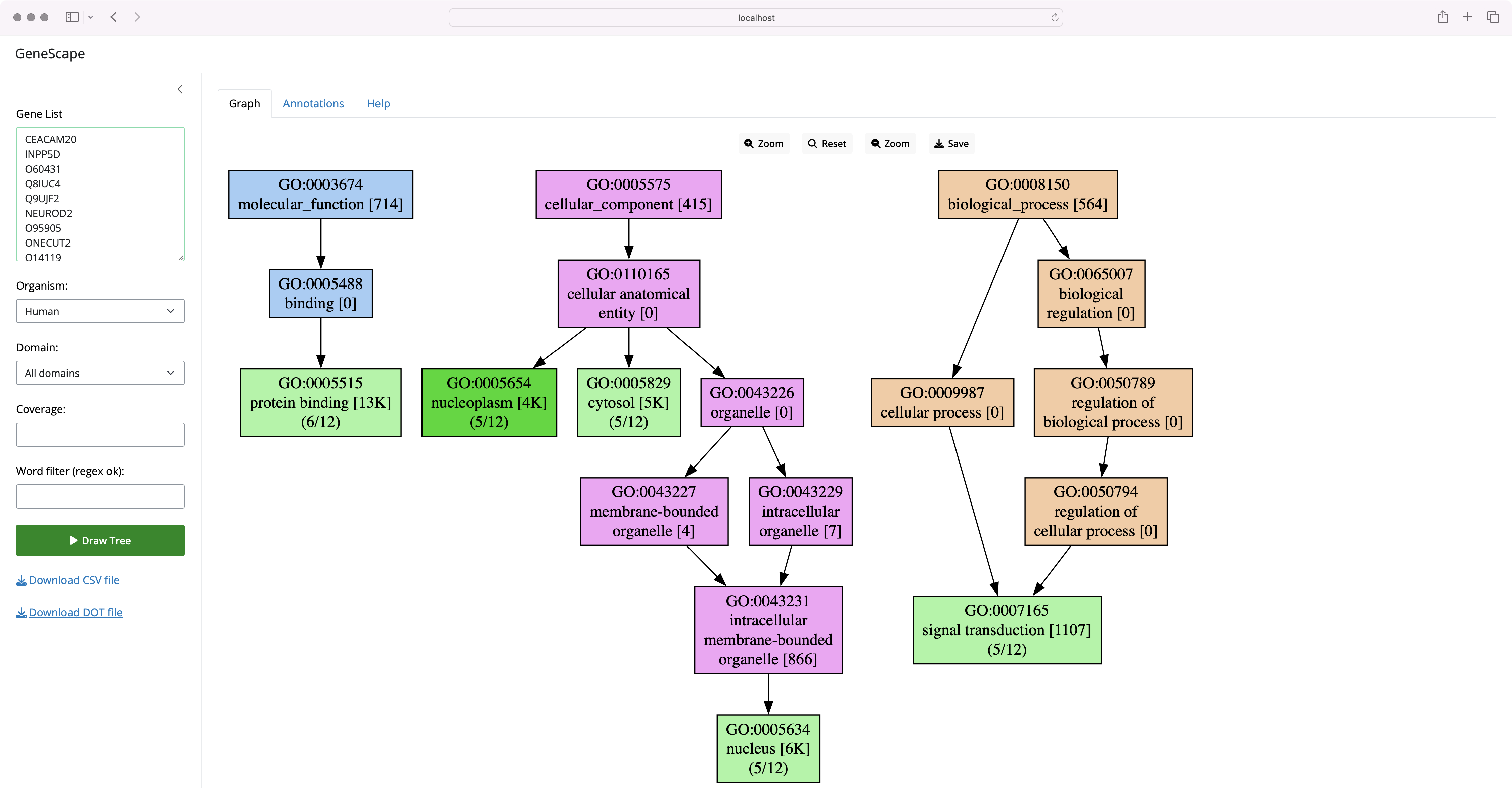
节点标签
图中的标签携带有关输入中具有该功能的基因数量的额外信息,并且是该功能在生物体中特异性的指示器。例如,标签
GO:0004866
endopeptidase
inhibitor activity [39]
(1/5)
标签表示功能内肽酶抑制剂活性被看作是原始关联文件中39个所有基因的注释(对于人类,有超过19K个基因符号)。因此,[39]是生物体注释的特征。
(1/5)表示输入列表中的5个基因中有1个具有此注释。因此,该值是输入列表的特征。将mincov(最小覆盖度)过滤器应用于覆盖度值以过滤出低于阈值的函数。
节点着色
树中的颜色具有额外的含义
- 浅绿色节点表示输入列表中的功能。
- 深绿色节点是输入中存在且在术语上是叶节点的功能,这是可能的最细粒度注释
深绿色表示该术语是叶节点,是可能的最具体注释。在这两种情况下,绿色表示功能存在于输入列表中。
不同GO类别的每个子树都有不同的颜色
- 生物学过程(BP)
- 分子功能(MF)
- 细胞组分(CC)
子树着色旨在帮助您了解可视化的功能术语的详细程度和特异性。
例如,1/4的数字表示输入中具有该功能的基因数量。
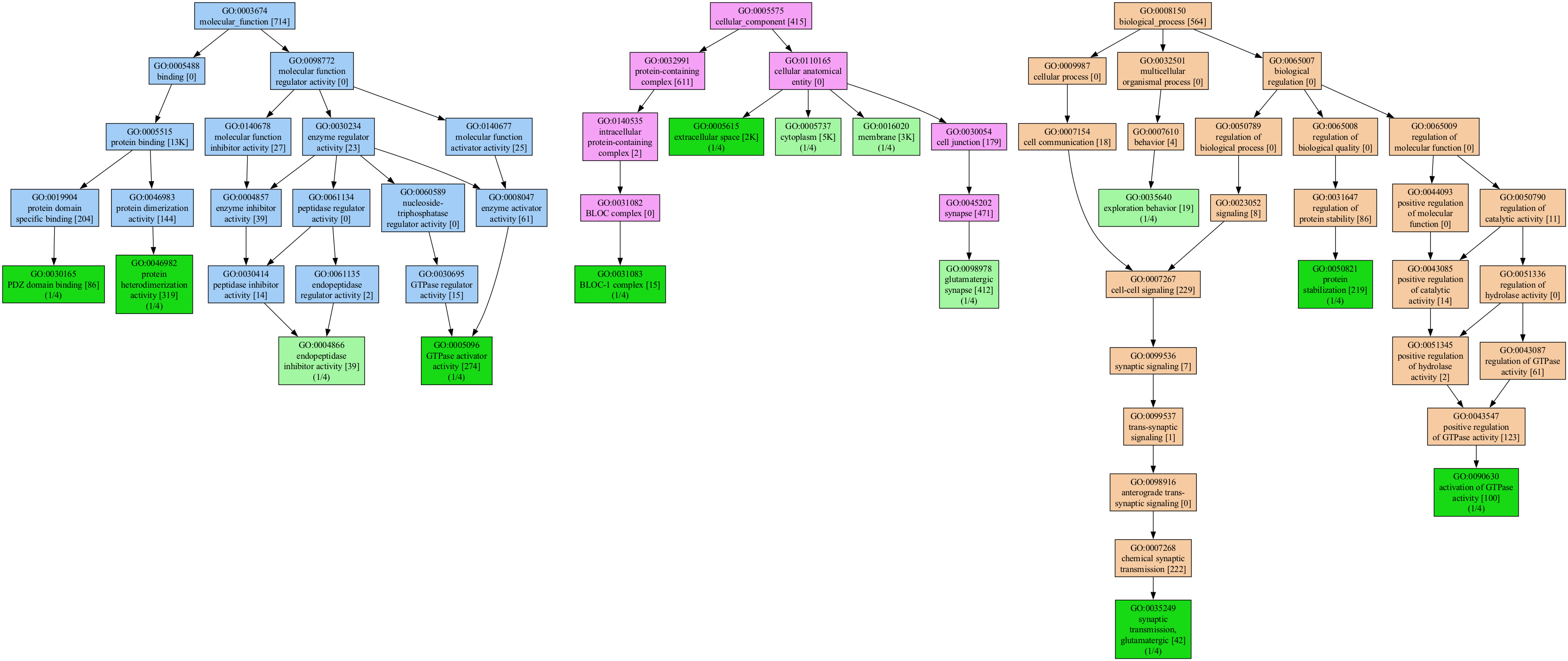
减少树的大小
即使基因数量很少,树也可能变得很大。
可以通过删除输入列表中不太代表性的功能或聚焦于仅包含匹配特定模式的功能来大大减少图形的大小。
将mincov设置为2或更高通常足以简化图形以使其易于管理。
用户可以应用的过滤条件包括
- 与功能列匹配的模式
- 最小覆盖度,表示具有该功能的基因的最小数量
- GO子树
过滤器在注释步骤中应用,并将过滤从基因列表中派生的GO术语。
在Shiny界面中,使用覆盖度过滤器删除输入列表中不太代表性的功能。回想一下,覆盖度代表输入列表中携带该功能的基因数量。您可以在“功能注释”框中看到每个注释的计数,作为第一列。
命令行要求
必须安装graphviz软件才能从命令行生成图像。您可以通过conda安装它
conda install graphviz
或通过apt或brew。
无法安装graphviz包的用户可以将输出保存为.dot文件
genescape tree --test -o output.dot
然后使用在线工具(如viz-js)来可视化图形。
genescape树
我们将测试数据打包到软件中,以便您可以像这样测试
genescape tree --test
这将生成测试数据的树形可视化。
减少图形大小
我们可以向树形可视化器传递基因列表或GO ID列表,甚至两者的混合列表。
我们运行tree命令来可视化包含所有覆盖率的GO术语之间的关系
genescape tree genes.txt --mincov 1
对于许多(大多数)基因列表,生成的功能图可能非常大。
如果未指定覆盖率,软件将尝试为输入基因找到一个合理的覆盖率阈值。
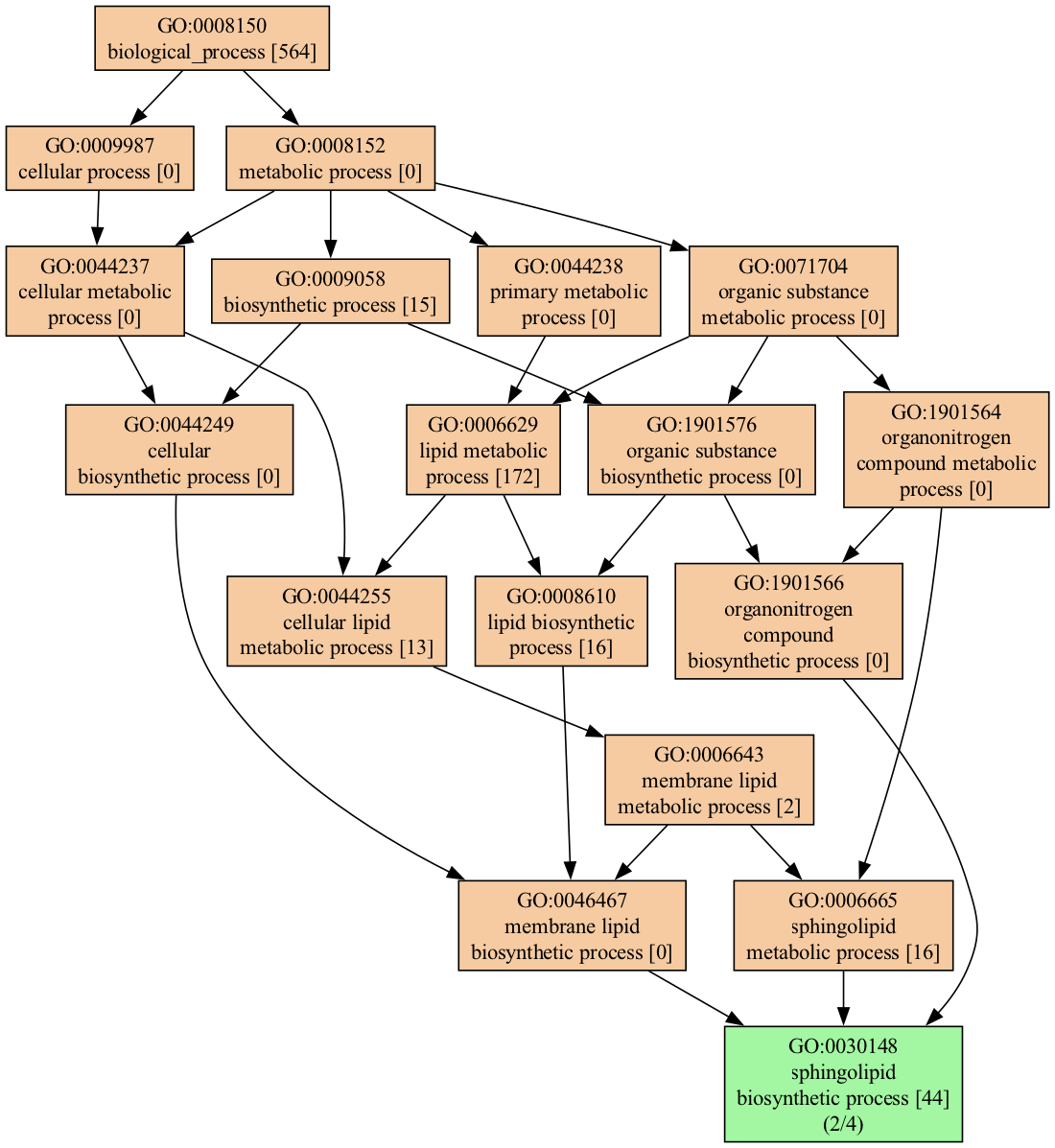
我们可以通过多种方式缩小可视化范围;例如,我们可以只选择匹配单词lipid的术语
genescape tree -m lipid --mincov 2 genes.txt
如上所示进行过滤后,输出结果更容易管理
genetrack注释
注释器作用于基因名。假设你有一份基因名列表,格式如下
Cyp1a1
Sphk2
Sptlc2
Smpd3
命令
genescape annotate genelist.txt
将产生以下输出
Coverage,Function,GO,Genes
3,protein binding,GO:0005515,CYP1A1|SMPD3|SPHK2
2,cytoplasm,GO:0005737,SMPD3|SPHK2
2,mitochondrial inner membrane,GO:0005743,CYP1A1|SPHK2
2,endoplasmic reticulum membrane,GO:0005789,CYP1A1|SPTLC2
2,sphingolipid biosynthetic process,GO:0030148,SPHK2|SPTLC2
2,intracellular membrane-bounded organelle,GO:0043231,CYP1A1|SPHK2
2,sphingosine biosynthetic process,GO:0046512,SPHK2|SPTLC2
genescape构建
该软件目前为多种生物体打包了索引。
要为不同的生物体构建索引,请从基因本体学网站下载GAF关联文件。
要构建新索引,请使用
genescape build --gaf mydata.gaf.gz --obo go.basic.gz -i mydata.index.gz
要使用自定义索引,将-i(--index)选项传递给任何web、tree和annotate命令,如下所示
genescape web --index mydata.index.gz
查看--help以获取更多选项。
其他事项
可以混合基因和本体学术语。以下是一个有效的输入
GO:0005488
GO:0005515
Cyp1a1
Sphk2
Sptlc2
测试
测试通过Makefile运行
make test
其他自定义
可以通过创建config.toml文件的副本并设置GENESCAPE_CONFIG环境变量指向新配置文件来自定义软件。
在此文件中,具有index类型的行将用于在Web界面中构建下拉菜单。
贡献
有关如何为GeneScape的开发做出贡献的信息,请参阅CONTRIBUTING.md。
引用
@article{GeneScape,
author = {Albert, Istvan},
doi = {10.21105/joss.06624},
journal = {Journal of Open Source Software},
month = jun,
number = {98},
pages = {6624},
title = {{GeneScape: A Python package for gene ontology visualization}},
url = {https://joss.theoj.org/papers/10.21105/joss.06624},
volume = {9},
year = {2024}
}
许可
genescape在MIT许可的条款下分发。
项目详情
下载文件
下载适合您平台文件的文件。如果您不确定选择哪个,请了解更多关于安装包的信息。