运行来自Blue Brain项目细胞包(如sscx、突触可塑性等)的细胞。
项目描述
EModelRunner
最新发布 |
|
文档 |
|
许可证 |
|
构建状态 |
|
覆盖率 |
|
支持 |
|
引用 |
介绍
EModelRunner是一个Python库,旨在以简单直接的方式运行Blue Brain门户提供的细胞模型。
它包含两个主要模块
一个模块用于展示突触可塑性现象。它具有以下功能
一个模块,用于运行来自SomatoSensoryCortex门户的细胞,具有以下功能
能够使用步骤和斜坡协议
突触刺激
能够生成hoc文件以运行细胞
图形用户界面
请注意,EModelRunner是一个便利工具,并不旨在成为通用的模拟器。
引用
当您使用此EModelRunner软件进行研究时,我们要求您引用以下出版物(这包括海报展示)
@article{emodelrunner,
title={EModelRunner},
DOI={10.5281/zenodo.8116075},
url={https://doi.org/10.5281/zenodo.8116075}
abstractNote={EModelRunner is a python library designed to run the cell models provided by the Blue Brain portals in a simple and straightforward way. It consists of two main modules. The first module is designed to show the synapse plasticity phenomenonand to run the cells and reproduce the results from the paper 'A calcium-based plasticity model for predicting long-term potentiation and depression in the neocortex'. The second module is designed to run the cells from the Blue Brain Project SomatoSensoryCortex portal. Note that EModelRunner is a convenience tool and does not aim to be a universal simulator.},
publisher={Zenodo},
author={Jaquier, Aurélien and Tuncel, Anil and Van Geit, Werner},
year={2023},
month={Jul}
}支持
我们提供Gitter上的聊天通道支持。
安装EModelRunner
安装EModelRunner的常用方法是使用pip。在这种情况下,您可能希望使用python虚拟环境。
使用pip安装
pip install emodelrunner
从源代码安装
如果您想修改emodelrunner,您可能想使用源代码仓库安装它。上述部分的说明同样适用,唯一的区别是您需要克隆git仓库
git clone https://github.com/BlueBrain/EModelRunner.git
并在新创建的emodelrunner子目录中运行pip(不要忘记命令末尾的点)
pip install -e .
支持的系统
emodelrunner的代码可以安装在支持pip安装Python代码的任何POSIX系统上。
依赖项
EModelRunner的主要依赖项包括
Python3.8+ Matplotlib Numpy Neurom H5py NEURON BluePyOpt Schema
理想情况下,遵循这些工具的安装说明,或使用预先安装的版本。
Python
现代Linux系统将已安装Python。请确保您的Python版本为3.8或更高。
请确保您使用的是最新的pip版本。最好在安装任何其他东西之前运行
pip install pip --upgrade
。
示例
突触可塑性示例
您可以在example/synplas_sample_dir中找到一个示例细胞包。转到此文件夹,或转到已设计用于与突触可塑性函数一起工作的细胞包。
运行模拟
运行模拟应该像这样简单
sh run.sh config_path
其中config_path是特定配置文件的路径。您将在config文件夹中找到可用的配置文件。它将使用预定义的突触前细胞尖峰训练来刺激突触运行突触后细胞。
您也可以进行完整的配对模拟,其中同时模拟突触前和突触后细胞。这应该像这样简单
sh run_pairsim.sh config_path
其中config_path是特定配置文件的路径。您将在config文件夹中找到可用的配置文件。
一旦模拟完成,输出将存储为output_{protocol_details}.h5。如果也模拟了预细胞,则其输出存储为output_precell_{protocol_details}.h5。
请注意,由于很难使突触前细胞在预先录制的尖峰训练文件中精确地同时发生尖峰(特别是当突触前细胞需要连续多次发生尖峰时),“完整配对”模拟的结果可能与“仅突触后细胞”模拟的结果略有不同。
所有配置文件都适用于“仅突触后细胞”和“完整配对”模拟。
分析输出
首先,使用以下命令加载有用的分析类
from emodelrunner.synplas_analysis import Experiment
然后创建一个Experiment实例
exp = Experiment(data="output_50Hz_10ms.h5", c01duration=, c02duration=, period=)
数据是输出文件的路径,或者包含输出数据的字典,分别用c01duration和c02duration表示EPSP监测在诱导方案前后的持续时间,周期是刺激突触的周期。
注意,如果诱导方案前后刺激突触所用的周期不同,可以将周期参数替换为c01period和c02period。
然后可以通过使用compute_epsp_ratio方法来计算EPSP的变化,如下所示
EPSP_ratio = exp.compute_epsp_ratio(n=60, method="amplitude)
其中n是要考虑的平均EPSP计算的扫描次数,method是计算EPSP比率的算法(可以是“幅度”或“斜率”)。
Sscx示例
您可以在example/sscx_sample_dir中找到一个示例细胞包。进入此文件夹,或者进入一个已设计用于与sscx函数一起工作的细胞包。
使用Python运行模拟
运行模拟应该像这样简单
sh run.sh config_path
其中config_path是特定配置文件的路径。您可以在config文件夹中找到可用的配置文件。请注意,所使用的方案将取决于配置文件的内容。
输出可以在python_recordings下找到。
使用hoc运行模拟
您还可以使用hoc运行模拟。为此,您必须首先使用以下行创建hoc文件
python -m emodelrunner.create_hoc --config_path config_path
其中config_path是特定配置文件的路径。您可以在config文件夹中找到可用的配置文件。请注意,配置文件中并非所有方案都可以转换为hoc。
然后使用以下命令运行模拟
sh run_hoc.sh
输出可以在python_recordings下找到。
GUI
sscx细胞也有一个GUI。要启动它,您必须进入一个兼容sscx的细胞包,然后输入
python -m emodelrunner.GUI --config_path config_path
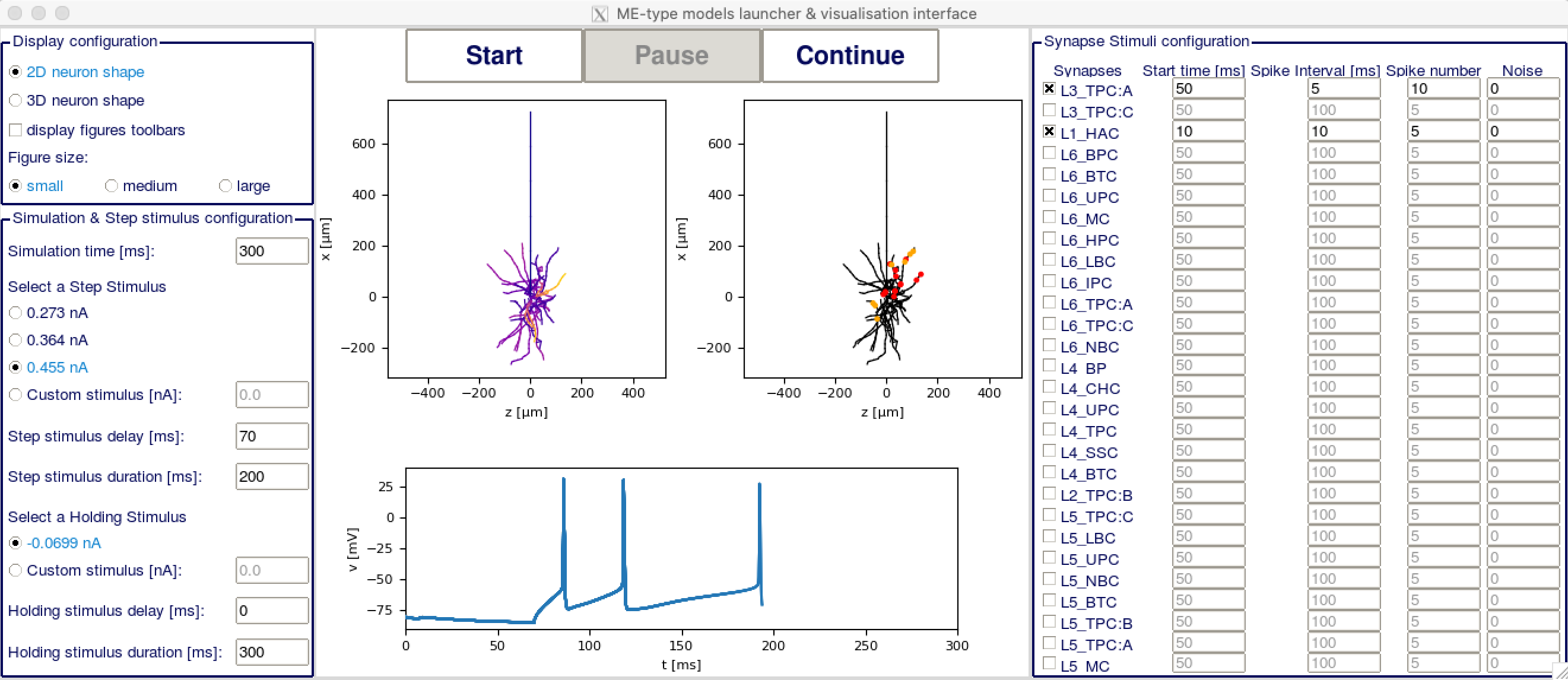
GUI的使用方法基本上是自我解释的。
在左列的上部,您有显示配置。您可能需要根据您的屏幕尺寸更改图形大小以实现最佳显示。在左列的下部是步进和保持刺激配置。您可以设置它们为自定义刺激,如果不需要任何步进刺激,则将它们设置为0。
在右列中,您有突触刺激配置。勾选您想要接收刺激的每个突触mtype的复选框。激活的突触将显示在右侧的图形中,兴奋性突触用红色点表示,抑制性突触用黄色点表示。然后您可以在右侧列中设置每个突触组何时开始放电,放电间隔和放电次数,以及是否应该有任何噪声。
在GUI的中心部分,有两个细胞图,左边的图显示每个部分的电压,右边的图显示突触位置。您可以用鼠标旋转这两个图的3D旋转。下面是显示随时间变化的树突电压的图。在顶部有三个按钮可以(重新)启动模拟,暂停它或恢复它。
资助与致谢
该项目的发展得到了瑞士联邦理工学院(EPFL)的洛桑联邦理工学院(EPFL)的一个研究机构——蓝色大脑项目的资金支持。
版权(c)2020-2024蓝色大脑项目/EPFL
项目详情
下载文件
下载适用于您的平台文件。如果您不确定选择哪个,请了解有关安装包的更多信息。
源分发
构建分发
emodelrunner-1.2.5.tar.gz的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 41e7a5efdc57726f168c7eef39eee78bde661cc9e37e666f2483c4f255d0de3c |
|
| MD5 | 45aa4f36f3ef35e2407939840ee59c9c |
|
| BLAKE2b-256 | b75c54773bec8d785d65be144fc366c7329b4904db2324881900d9d835cf918d |
EModelRunner-1.2.5-py3-none-any.whl的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 018f7a41ca597eed28c29caecab99f6ae3a56c825554409d996526af7f6543bd |
|
| MD5 | c5f8106fe36a09891dd989d262e5141b |
|
| BLAKE2b-256 | 77a03f441612a59d9cac203e47ab252857b791fce6fe3038b34351eae2328d84 |