DeepLC:使用深度学习进行(修改后)肽的保留时间预测。
项目描述

DeepLC:使用深度学习进行(修改后)肽的保留时间预测。
简介
DeepLC是一种采用深度学习的(修饰)肽保留时间预测器。其优势在于,即使训练过程中未见过该修饰,也能准确预测修饰肽的保留时间。
DeepLC可以通过网络应用、带有图形用户界面(GUI)的本地使用,或作为Python包使用。在后一种情况下,DeepLC可以通过命令行或作为Python模块使用。
引用
如果您在研究中使用DeepLC,请使用以下引用
DeepLC可以预测携带尚未见过的修饰的肽的保留时间
Robbin Bouwmeester,Ralf Gabriels,Niels Hulstaert,Lennart Martens & Sven Degroeve
Nature Methods 18, 1363–1369 (2021) doi: 10.1038/s41592-021-01301-5
用法
Web应用程序
只需访问iomics.ugent.be/deeplc即可开始使用!
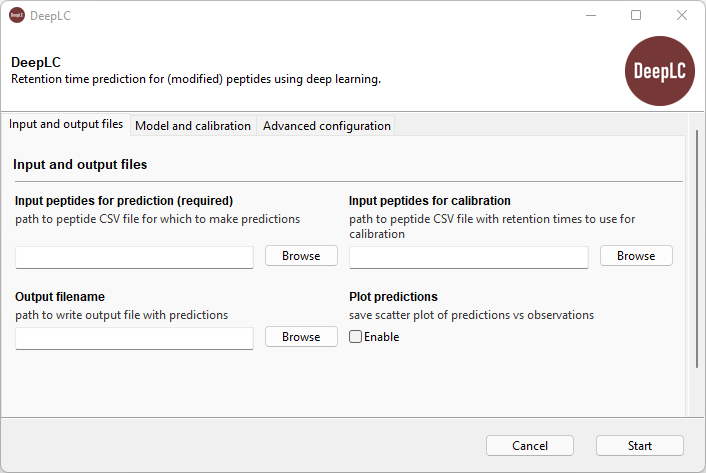
图形用户界面
在现有的Python环境中(跨平台)
- 在安装了Python(>=3.7)的终端中,运行
pip install deeplc[gui] - 使用命令
deeplc-gui或python -m deeplc.gui启动GUI
独立安装程序(Windows)
- 从最新版本下载DeepLC安装程序(
DeepLC-...-Windows-64bit.exe) - 执行安装程序
- 如果Windows Smartscreen显示带有“Windows保护了您的PC”的弹出窗口,请单击“更多信息”然后单击“仍然运行”。您需要信任DeepLC不包含任何病毒,或者您可以检查源代码😉
- 按照安装步骤进行
- 启动DeepLC!
Python包
安装
使用conda安装,使用bioconda和conda-forge通道:conda install -c bioconda -c conda-forge deeplc
或使用pip安装:pip install deeplc
命令行界面
要使用DeepLC CLI,请运行
deeplc --file_pred <path/to/peptide_file.csv>
我们强烈建议添加一个已知保留时间的肽文件以进行校准
deeplc --file_pred <path/to/peptide_file.csv> --file_cal <path/to/peptide_file_with_tr.csv>
有关所有CLI参数的概述,请运行deeplc --help。
Python模块
最小示例
import pandas as pd
from deeplc import DeepLC
peptide_file = "datasets/test_pred.csv"
calibration_file = "datasets/test_train.csv"
pep_df = pd.read_csv(peptide_file, sep=",")
pep_df['modifications'] = pep_df['modifications'].fillna("")
cal_df = pd.read_csv(calibration_file, sep=",")
cal_df['modifications'] = cal_df['modifications'].fillna("")
dlc = DeepLC()
dlc.calibrate_preds(seq_df=cal_df)
preds = dlc.make_preds(seq_df=pep_df)
有关更详细的示例,请参阅examples/deeplc_example.py .
输入文件
DeepLC期望的是逗号分隔的值(CSV),以下列出了以下列
seq:未修饰的肽序列modifications:MS2PIP风格格式的修饰:每个修饰都列为例location|name,位置、名称和其他修饰之间用竖线(|)分隔。location是一个整数,从第一个氨基酸开始计数,1。0保留用于N端修饰,-1用于C端修饰。name必须与Unimod(PSI-MS)名称相对应。tr:保留时间(仅在校准时需要)
例如
seq,modifications,tr
AAGPSLSHTSGGTQSK,,12.1645
AAINQKLIETGER,6|Acetyl,34.095
AANDAGYFNDEMAPIEVKTK,12|Oxidation|18|Acetyl,37.3765
有关更多示例,请参阅examples/datasets。
预测模型
DeepLC包含多个在来自各种实验设置的数据上训练的CNN模型。默认情况下,DeepLC根据校准数据集选择最佳模型。如果没有执行校准,则选择第一个默认模型。始终注意使用的模型和DeepLC版本。当前版本包含
| 模型文件名 | 实验设置 | 出版物 |
|---|---|---|
| full_hc_PXD005573_mcp_8c22d89667368f2f02ad996469ba157e.hdf5 | 反相色谱 | Bruderer等人2017年 |
| full_hc_PXD005573_mcp_1fd8363d9af9dcad3be7553c39396960.hdf5 | 反相色谱 | Bruderer等人2017年 |
| full_hc_PXD005573_mcp_cb975cfdd4105f97efa0b3afffe075cc.hdf5 | 反相色谱 | Bruderer等人2017年 |
有关DeepLC中可以使用的所有完整模型(包括一些TMT模型!),请参阅
https://github.com/RobbinBouwmeester/DeepLCModels
模型的命名规范如下
[full_hc]_[dataset]_[fixed_mods]_[hash].hdf5
不同的部分表示
full_hc - 标志表示已完成、训练和完全优化的模型
dataset - 用于拟合模型的数据集名称(参见原始出版物,补充表2)
fixed mods - 标志表示已添加到肽中,但没有明确指示的固定修饰(例如,半胱氨酸的羰甲基化)
哈希 - 表示不同的架构,其中 "1fd8363d9af9dcad3be7553c39396960" 表示 CNN 滤波器长度为 8,"cb975cfdd4105f97efa0b3afffe075cc" 表示 CNN 滤波器长度为 4,而 "8c22d89667368f2f02ad996469ba157e" 表示滤波器长度为 2。
问答
问题:是否需要在输入文件中指示固定的修改?
是的,即使是像氨基甲酰甲基这样的修改也应该在输入文件中。
问题:所以 DeepLC 能够预测任何修改的保留时间吗?
是的,DeepLC 可以预测任何修改的保留时间。然而,如果修改与模型在训练期间看到的肽序列差异很大,预测的准确性可能不会令人满意。例如,如果模型从未见过磷酸原子,预测的准确性将会很低。
问题:安装失败。为什么?
请确保将 DeepLC 安装在一个不包含空格的路径中。运行最新的 LTS 版本的 Ubuntu 或 Windows 10。请确保你有足够的磁盘空间,出人意料的是,TensorFlow 需要相当多的磁盘空间。如果你仍然无法安装 DeepLC,请随时联系我们。
Robbin.Bouwmeester@ugent.be 和 Ralf.Gabriels@ugent.be
问题:我有不支持的特殊用例。你能帮帮我吗?
当然,请随时联系我们。
Robbin.Bouwmeester@ugent.be 和 Ralf.Gabriels@ugent.be
问题:DeepLC 耗尽内存。我该怎么做?
你可以尝试减小批处理大小。如果批处理大小足够低,即使是在只有 4 GB RAM 的机器上,DeepLC 也应该能够运行。
问题:我有一块显卡,但 DeepLC 没有使用 GPU。为什么?
目前 DeepLC 默认使用 CPU 而不是 GPU。显然,因为你想要使用 GPU,你是一位高级用户 :-)。如果你想最大限度地利用昂贵的 GPU,你需要更改或删除 deeplc.py(在顶部)中的以下行
# Set to force CPU calculations
os.environ['CUDA_VISIBLE_DEVICES'] = '-1'
还要在 reset_keras() 函数中更改相同的行
# Set to force CPU calculations
os.environ['CUDA_VISIBLE_DEVICES'] = '-1'
可以删除该行或将其更改为(其中数字表示 GPU 的数量)
# Set to force CPU calculations
os.environ['CUDA_VISIBLE_DEVICES'] = '1'
问题:我应该使用什么修改名称?
使用 unimod 的名称。默认情况下使用 PSI-MS 名称,但如果 PSI-MS 名称不可用,则使用临时名称作为备用。请参阅 unimod/ 文件夹中的 unimod_to_formula.csv 以了解特定修改的命名。
问题:我有不在 unimod 中的修改。我该如何添加修改?
在 unimod/ 文件夹中有一个名为 unimod_to_formula.csv 的文件,可用于添加修改。在 CSV 文件中添加一个名称(必须是唯一的且尚未存在)以及原子组成的改变。例如
Met->Hse,O,H(-2) C(-1) S(-1)
请确保使用负号表示减去的原子。
问题:帮助,我的所有预测都在 [0,10] 之间。为什么?
很可能是你没有使用校准。没问题,但用于训练的保留时间在 [0,10] 之间进行了归一化。这意味着你可能需要在分析后自己调整保留时间,或者使用校准集作为输入。
问题:选项 dict_divider 是做什么的?
此参数定义了用于校准保留时间快速查找的精度。值为 10 表示精度为 0.1(值为 100 表示精度为 0.01)在校准锚点之间。此参数不会影响校准的精度,但将其设置得太高可能意味着在锚点之间模型选择不当。通常,一个安全值是大于 10 的。
问题:选项 split_cal 是做什么的?
选项 split_cal,或称为分段校准,用于设置色谱图进行分段线性校准的分割数量。如果将值设置为10,则色谱图将分成10个等距的部分。对于每个部分,选择校准肽段的中间值。这些是锚点。在每一个锚点之间进行线性拟合。当使用pyGAM广义加性模型进行校准时,此选项不起作用。
问:DeepLC的集成部分是如何工作的?
如果模型名称重叠,则同一目录下的模型将被分组。重叠必须出现在名称的全名中,但名称中“_”字符之后的部分除外。
以下模型将被分组
full_hc_dia_fixed_mods_a.hdf5
full_hc_dia_fixed_mods_b.hdf5
以下模型将不会被分组
full_hc_dia_fixed_mods2_a.hdf5
full_hc_dia_fixed_mods_b.hdf5
full_hc_dia_fixed_mods_2_b.hdf5
问:我想对多个模型进行集成平均,即使它们在不同的数据集上训练。我该如何操作?
请随意实验!如果模型名称重叠,则同一目录下的模型将被分组。重叠必须出现在名称的全名中,但名称中“_”字符之后的部分除外。
以下模型将被分组
model_dataset1.hdf5
model_dataset2.hdf5
因此,您只需要重命名您的模型。
项目详情
下载文件
下载适用于您平台的文件。如果您不确定要选择哪个,请了解有关 安装包 的更多信息。
源代码分发
构建分发
deeplc-3.1.0.tar.gz的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 584b2dbda0b9c2a7ef7dd413cc228387a7ddc5813d556e929be6ef7c54e0d679 |
|
| MD5 | 6bc4c58d009e84b7d313050a251b3648 |
|
| BLAKE2b-256 | 2ccbcd6dc6d6207edd3685646e37129a17fff1b385e6a2584d3b58fafcb67434 |
deeplc-3.1.0-py3-none-any.whl的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 923124de8a31330b5263833bba44115290eec8b4ca08fdc34869b3fb0f571b9b |
|
| MD5 | e2d34c91d1b7c95c6066342b426a0462 |
|
| BLAKE2b-256 | f0b77cbfd0be88ba3952a4994ed3736a98796c9a2abff75aa1d61c6132ff4d35 |