一个多功能的工具,用于对.cool格式的Hi-C数据进行堆叠分析。
项目描述
coolpup.py
.cool文件使用
一个多功能的工具,用于对.cool格式的Hi-C数据进行堆叠分析(https://github.com/mirnylab/cooler)。谁不喜欢可爱的小狗呢?
介绍
什么是堆叠?
堆叠是我们用来描述任何平均多个2D矩阵(片段)的2D区域(过程)的通用术语,例如Hi-C数据。在某些情况下,它们也被称为APA(聚合峰分析,来自Rao等,2014年),或聚合区域/TAD分析(在GENOVA中,van der Weide等,2021年),以及其他名称。最典型的用例是量化Hi-C数据中调用点的(环)的平均强度或TAD边界的强度。然而,这种方法可以做得更多。这是检查某些区域是否倾向于相互作用的堆叠工作原理。
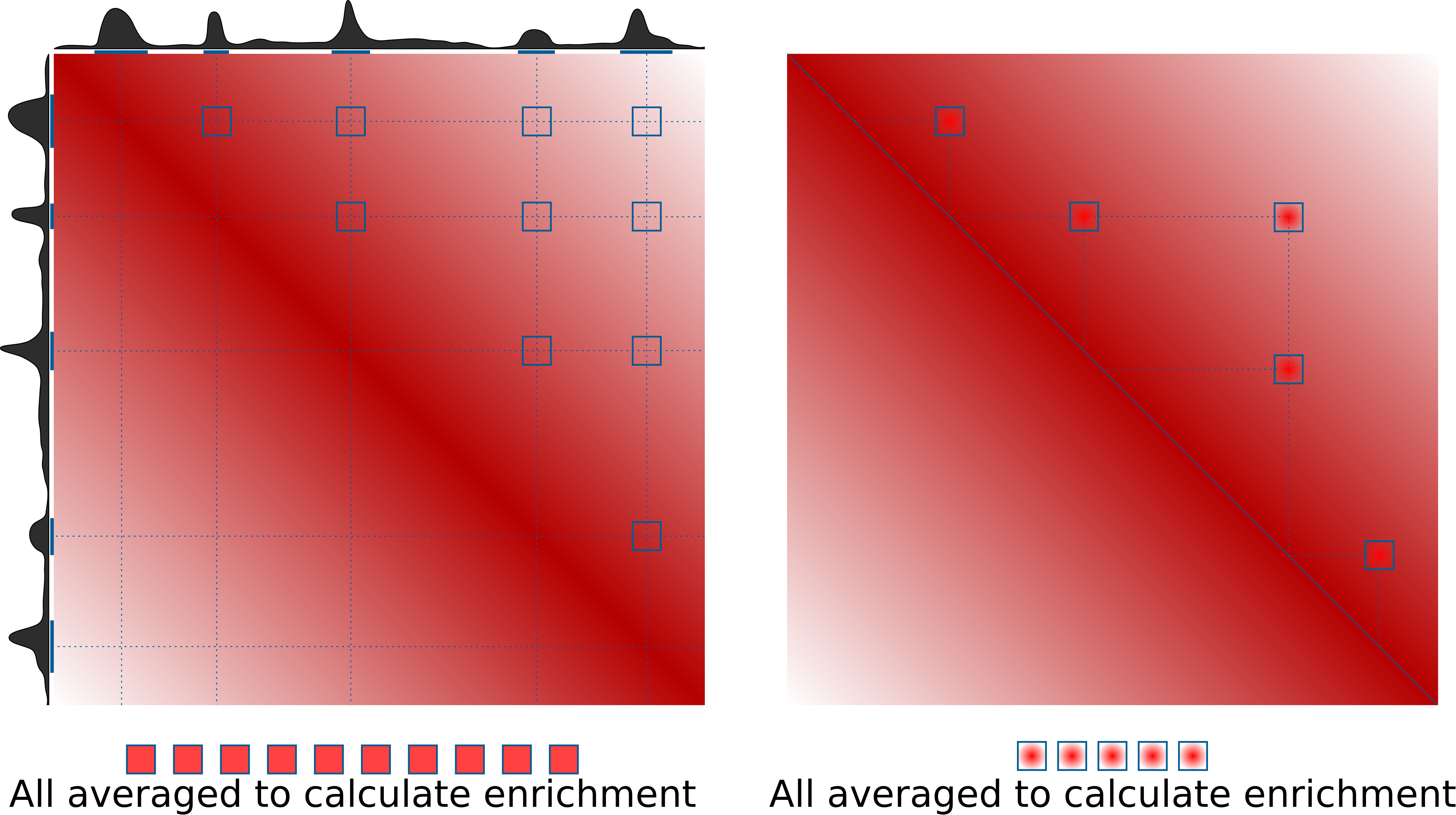
在右侧是量化环强度的更典型用例。在左侧是不同的方法,旨在检查基因组中特定区域(例如,特定因子的结合位点)是否倾向于相互作用。
对于这个量化来说,非常重要的步骤是将数据归一化到预期值。这可以通过两种方式实现:一种是通过使用染色体(或臂)范围的距离预期相互作用,使用包含不同距离间相互作用平均值(例如,cooltools expected-cis的输出)的文件;另一种方式是直接通过将随机移动的控制区域中的重叠分值进行除法从Hi-C数据中获取。如果不使用预期归一化方法(仅设置--nshifts 0),这基本上等同于APA方法(Rao等人,2014年),可用于平均强相互作用区域,例如注释的环。对于较弱的相互作用者,随着距离的增加接触概率的衰减可能会隐藏任何可能观察到的局部富集。然而,最重要的是,在比较不同距离的甚至略微不同的区域集或比较不同数据集时,距离与接触概率的衰减将非常强烈地影响结果值,因此在很多情况下对它进行归一化是必要的,并且通常推荐。
coolpup.py与cooltools pileup
cooltools是由open2C维护的用于Hi-C分析的主要包,它还包含一个进行重叠分值的工具。那么为什么存在coolpup.py呢?cooltools pileup的工作方式是,它将所有重叠分值的片段累积到一个3D数组(堆栈)中。这为后来基于某些特征对片段进行子集划分或基于堆栈进行其他非标准计算提供了很大的灵活性。但这只有在使用Python API进行分析时才具有优势,并且还限制了cooltools pileup的应用,因为它由于内存需求无法应用于大量片段。这就是coolpup.py的用武之地:在内部,它从不将超过一个片段存储在内存中,因此可以处理任意数量的片段。由于它逐个将整个染色体加载到内存中(或并行加载以加速处理),因此coolpup.py在分析大量潜在相互作用方面表现优异,因为它可以快速提取小子矩阵。读取所有内容到内存使得在处理少量环时相对较慢,但性能下降直到达到大量相互作用。
尽管在生成重叠分值后无法对片段进行子集划分(因为它们没有被存储),但coolpup.py允许在重叠分值过程中进行各种子集划分。CLI中的内置选项包括按距离、按链、同时按链和距离以及按窗口/区域进行子集划分 - 在提供BED文件的情况下,对于同一染色体中的每一行都生成一个针对所有其他行的重叠分值;在转置重叠分值的情况下,可以为每个染色体对生成重叠分值。重要的是,在Python API中可以执行任意片段组合的子集划分。
.cool格式
.cool是一种现代且灵活的格式,用于存储Hi-C数据。它使用HDF5存储Hi-C数据的稀疏表示,这使得处理高分辨率数据集时内存需求低。另一种流行的存储Hi-C数据的格式.hic可以通过hic2cool(https://github.com/4dn-dcic/hic2cool)转换为.cool文件。
详见
Abdennur, N.和Mirny, L.(2019). Cooler:用于Hi-C数据和其他基因组标记数组的可扩展存储。Bioinformatics。10.1093/bioinformatics/btz540
入门指南
安装
所有其他要求都可以从PyPI或conda获取。
在安装所有内容之前,您需要使用pip或conda获取Cython。然后对于coolpuppy(以及其他依赖项)只需执行以下操作:
pip install coolpuppy
或
pip install https://github.com/open2c/coolpuppy/archive/master.zip
从GitHub获取最新版本。这将使coolpup.py在您的终端中可调用,并在Python中以coolpuppy的形式导入。
用法
基本用法语法如下
coolpup.py [选项] coolfile.cool regionfile.bed
堆叠分析指南教程在这里(WIP):教程
命令行界面文档在这里:CLI文档
一些用于开始使用CLI界面的示例在这里:这里,Python API示例请参阅这里。
绘制结果
为了灵活绘图,我建议使用matplotlib或其他库。然而,此包中包含简单的绘图功能。只需运行plotpup.py并使用所需选项,并列出您想绘图的coolpup.py的所有输出文件。
引用coolpup.py
Ilya M Flyamer,Robert S Illingworth,Wendy A Bickmore(2020)。Coolpup.py:Hi-C数据的灵活堆叠分析。生物信息学,36,10,2980–2985。
https://academic.oup.com/bioinformatics/article/36/10/2980/5719023
doi: 10.1093/bioinformatics/btaa073
项目详情
下载文件
下载适用于您平台的应用程序。如果您不确定选择哪个,请了解有关安装包的更多信息。
源分发
构建分发
coolpuppy-1.1.0.tar.gz的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | b40a551cf29bc6b7b377ecd68bccbb5923cbe5cae4d0426673461f3b1ff12141 |
|
| MD5 | bc112754f3668df1db54f4f456191b4d |
|
| BLAKE2b-256 | df668610264268a3a79970a03ef731697ebac84455ee64a8fe8b6dc7be5d8ea4 |
coolpuppy-1.1.0-py3-none-any.whl的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 70d5af3451da6fe122f2349658d06e6ff5378e353208dc84bbfb37387e1bffd7 |
|
| MD5 | 80791c2d909954ee5c8de21a52a8ab76 |
|
| BLAKE2b-256 | 43669fc3c0e81ed41e8c6daba9fb4227cb9fee160a913d841f6a66915cf7f3b2 |

















