使用深度集成进行组合特征选择
项目描述
复合特征选择
NeurIPS 2022论文的官方代码 使用深度集成进行组合特征选择 ( Fergus Imrie, Alexander Norcliffe, Pietro Liò, Mihaela van der Schaar )
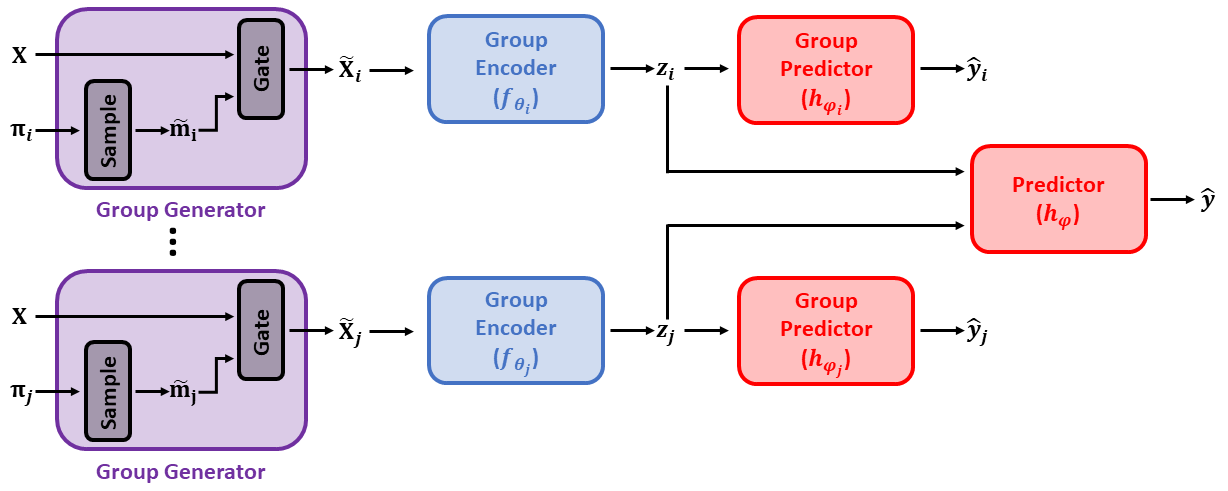
当前的特征选择方法仅返回预测特征的列表。然而,特征往往不会单独起作用,而是相互配合。以XOR作为简单的例子,特征1在不知道特征2的值的情况下实际上并不提供任何信息,反之亦然。这项工作旨在解决组合特征选择的问题,即找到一起工作的特征组。
摘要
在许多现实世界的问题中,特征并非单独起作用,而是相互结合。例如,在基因组学中,疾病可能不是由任何单个突变引起的,而是需要多个突变的存在。先前关于特征选择的研究要么试图识别单个特征,要么只能从预定义的集合中确定相关的组。我们研究了在不预先分组的情况下发现预测特征组的问题。为此,我们根据特征之间的线性和非线性交互定义了预测组。我们引入了一种新的深度学习架构,该架构使用特征选择模型的集成来找到预测组,而不需要提供候选组。选定的组是稀疏的,并且重叠最小。此外,我们提出了一种新的度量方法来衡量发现组与真实情况之间的相似度。我们在多个合成任务、半合成化学数据集以及已知真实结构图像数据集和现实世界癌症数据集上展示了我们模型的有用性。
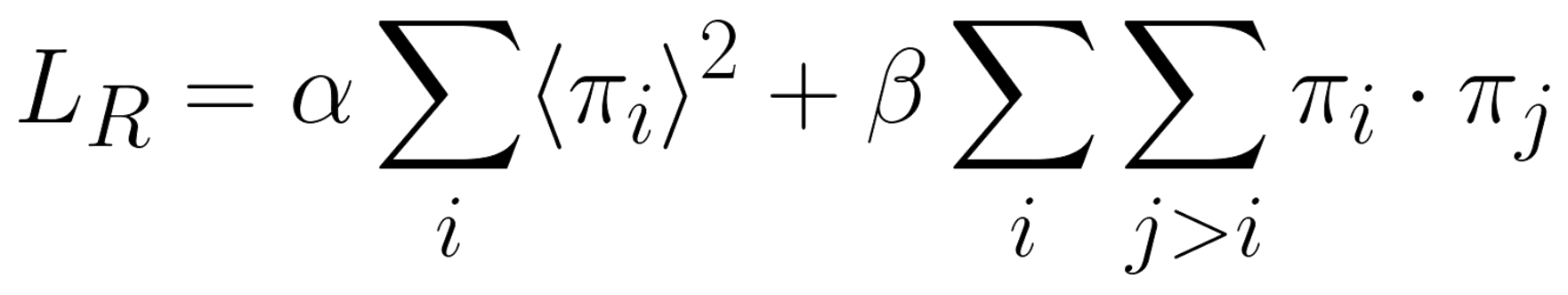
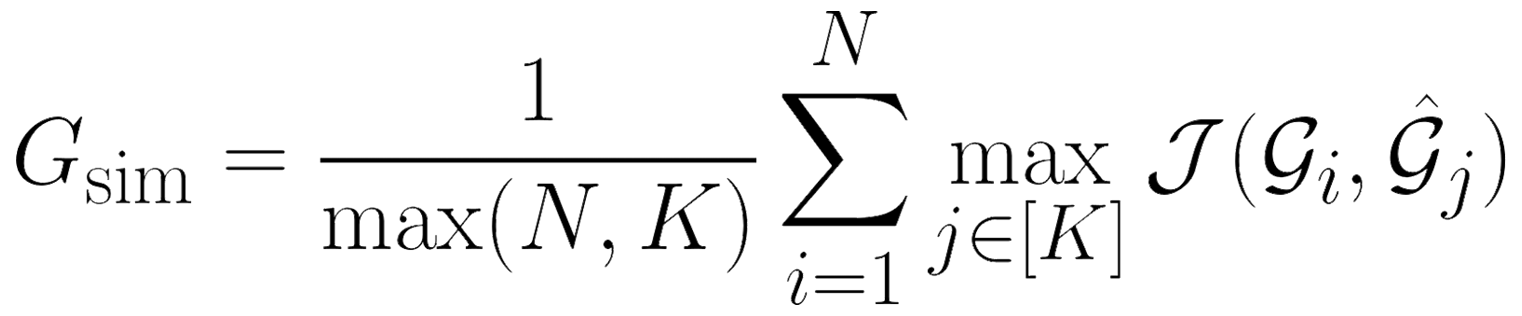
安装和使用
本项目使用了 python 3.7。要设置虚拟环境和必要的软件包,请运行以下命令
$ conda create --name compfs python=3.7
$ conda activate compfs
可以使用以下命令从PyPI安装库
pip install compfs
或从源安装,使用
pip install .
要运行测试和实验,可以使用以下命令安装依赖项
pip install .[testing]
数据集
为了节省空间,此处未包含数据集。请下载每个数据集并将其放置在以下文件夹中
- 化学数据: 链接,将数据存储在 <library_path>/datasets/chem_data/(从‘all_16_logics_train_and_test’文件夹复制粘贴)
- Metabric数据: 链接,将数据存储在 <library_path>/datasets/metabric_data/(从‘archive’文件夹复制粘贴)
运行实验
可以从命令行(从主目录)运行实验,参数为:实验编号、实验、模型,例如
$ python -m experiments.run_experiment --experiment_no 1 --experiment syn1 --model compfs1
$ python -m experiments.run_experiment --experiment_no 6 --experiment chem3 --model ensemble_stg
$ python -m experiments.run_experiment --experiment_no 2 --experiment metabric --model compfs
$ python -m experiments.run_experiment --experiment_no 1 --experiment mnist --model stg
之后,可以从命令行(从主目录)执行标准评估以生成论文中的表格,参数为:实验、模型,例如
$ python -m experiments.run_evaluation --experiment syn4 --model lasso
$ python -m experiments.run_evaluation --experiment chem1 --model random_forests
$ python -m experiments.run_evaluation --experiment chem2 --model oracle
对于Group Lasso、STG和Concrete Autoencoder,我们在experiments/notebooks/文件夹中包含了Python笔记本以供运行。还包括用于MNIST评估的笔记本。STG也可以使用相同的命令从命令行运行,命令行参数为--model stg,但是这是一个经过调整的集成STG,只有一个STG,因此已相应调整超参数以获得相同的积极结果。
在您的数据上运行CompFS
我们包含了一个名为'tutorials/example.ipynb'的笔记本,它展示了在Syn1上使用CompFS。在此,可以将数据轻松替换为自定义数据的NumPy数组,并设置CompFS的超参数。'standalone_example.ipynb'笔记本与该笔记本相同,但代码是直接复制的而不是导入的,因此可以不要求从存储库中导入即可使用。
引用
如果我们的论文或代码对您的自身研究有所帮助,请在您的文献中引用我们的工作如下
@article{imrie2022compfs,
title={{C}omposite {F}eature {S}election using {D}eep {E}nsembles},
author={Imrie, Fergus and Norcliffe, Alexander and Li{\`o}, Pietro and van der Schaar, Mihaela},
journal={Advances in Neural Information Processing Systems},
volume={35},
year={2022}
}
致谢
我们感谢匿名审稿人对本文的评论和建议。在本项工作期间,Fergus Imrie 和 Mihaela van der Schaar 受到美国国家科学基金会(NSF,项目编号 1722516)的支持。Mihaela van der Schaar 还得到海军研究办公室(ONR)的支持。Alexander Norcliffe 得到葛兰素史克(GlaxoSmithKline)的资助。我们还要感谢 Bogdan Cebere 和 Evgeny Saveliev 对此代码的审查。
项目详情
下载文件
下载适用于您平台的文件。如果您不确定选择哪个,请了解更多关于 安装包 的信息。
源代码分发
构建分发
哈希值 for compfs-0.0.2-py3-none-macosx_10_14_x86_64.whl
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | a68949009665cdb10e5c44a41edeba167d97fca0375bfdec808b60809962d9c6 |
|
| MD5 | 5dc5c0b9352488dc10f60b597ff7fecd |
|
| BLAKE2b-256 | bd41207b465ff9fa62dc29d52f430b99438c0b5676f446c4d88c902f7de7b817 |









