Blue Brain Python电生理建模管道
项目描述
BluePyEModel:Blue Brain Python电生理建模管道
最新发布版 |
|
文档 |
|
许可 |
|
构建状态 |
|
覆盖率 |
|
引用 |
介绍
Blue Brain Python电生理建模管道(BluePyEModel)是一个Python包,它简化了电生理神经元模型(e模型)构建任务的配置和执行。它涵盖了从电生理数据中提取电生理特征、e模型参数优化和模型验证等任务。因此,它建立在eFEL、BluePyEfe和BluePyOpt的基础上。
关于电气模型构建的一般概述和示例,请参阅以下论文:[一个创建、验证和泛化详细神经元模型的通用工作流程](https://doi.org/10.1016/j.patter.2023.100855)。
请注意,本包仅涵盖基于电生理数据(patch-clamp)的电气模型构建,且仅依赖于NEURON模拟器。
引用方式
如果您在工作中使用了此存储库,请参阅存储库页面顶部的引用此存储库按钮以获取各种引用格式,包括APA和BibTeX。
有关详细的引用信息,请参阅CITATION.cff文件。
安装
可以使用以下命令通过pip安装BluePyEModel:
pip install bluepyemodel[all]如果您不想安装所有依赖项,可以通过在括号中指定要安装的依赖项来选择特定的依赖项,而不是使用“all”。如果需要多个依赖项,它们必须由逗号分隔。可用的依赖项有:
luigi
nexus
all
开始E-模型构建流程
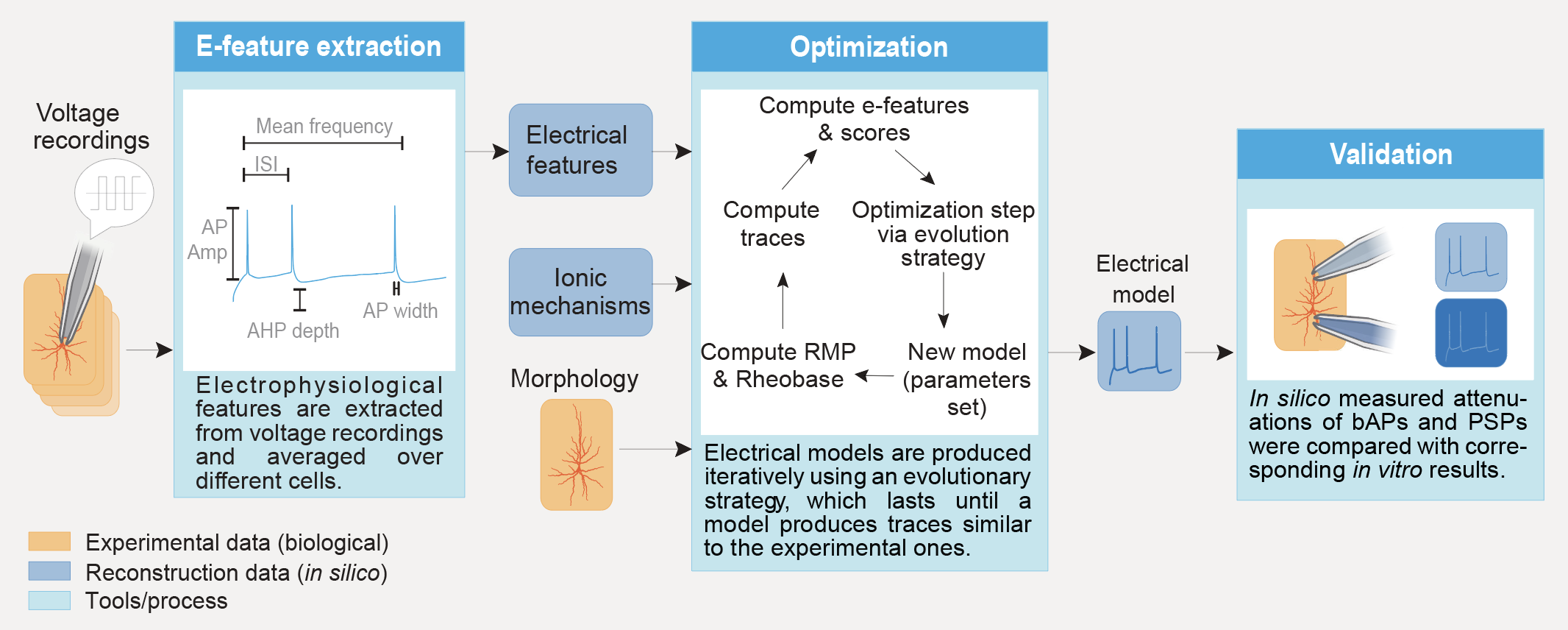
本节展示了流程的一般图景。有关详细图景和使用方法,请参阅L5PC示例及其README。
流程分为6个步骤
提取:从电生理记录中提取e特征,并沿请求的目标平均e特征值。
优化:构建NEURON细胞模型,并使用在e特征提取期间计算的e特征作为目标来优化其参数。
存储模型:读取提取结果并将模型(最佳参数集)存储在本地json文件中。
验证:读取模型并在其上运行优化协议和/或验证协议。然后,将在此协议上获得的e特征得分传递给验证函数,以决定模型是否足够好。
绘图:读取模型并在其上运行优化协议和/或验证协议。然后,沿e特征得分和参数分布绘制结果轨迹。
导出:读取最佳模型的参数并将它们导出到可以在NEURON中使用的文件或用于电路构建的文件中。
这六个步骤应按顺序运行,例如,如果没有存储模型,则无法运行验证。步骤验证、绘图和导出是可选的。在手动创建或从旧项目获取协议和优化目标文件的情况下,步骤提取也可以是可选的。
有关如何使用Nexus访问点运行流程的示例,请参阅Nexus示例
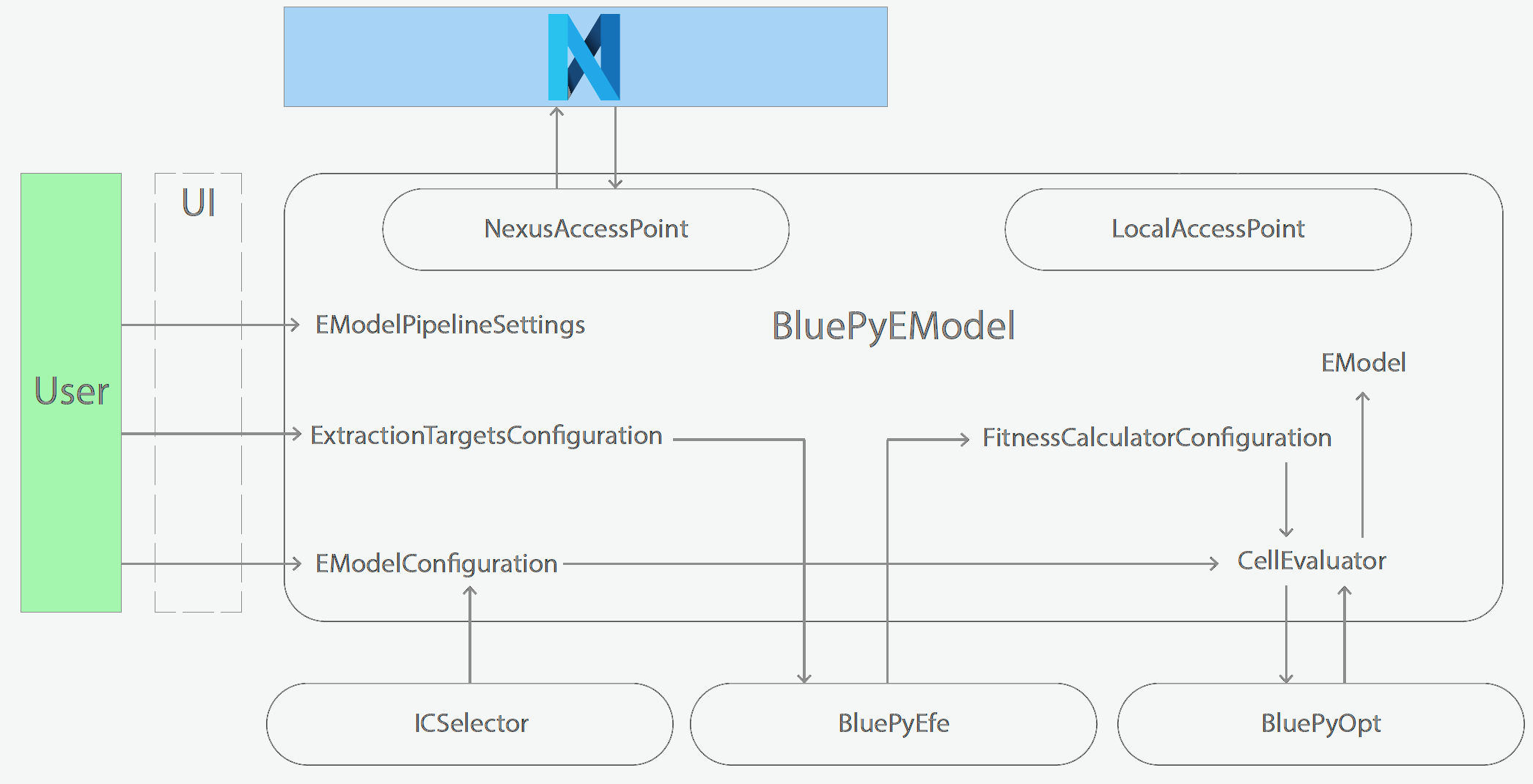
BluePyEModel类图
致谢
这项工作得到了瑞士联邦理工学院(EPFL)洛桑联邦理工学院(EPFL)的研究中心——蓝脑计划资助,该计划由瑞士联邦政府的技术学院委员会资助。本工作部分资助由欧盟第七框架计划(FP7/2007-2013)根据协议号604102(HBP)提供,并由欧盟的“地平线2020”研究与创新框架计划根据特定资助协议号720270(人脑项目SGA1)和785907(人脑项目SGA2)提供,以及由EBRAINS研究基础设施提供,该基础设施由欧盟“地平线2020”研究与创新框架计划根据特定资助协议号945539(人脑项目SGA3)提供资金。
版权
版权(c)2023-2024 蓝脑计划/EPFL
本作品根据Apache 2.0授权
项目详情
下载文件
下载适合您平台的文件。如果您不确定选择哪个,请了解有关安装包的更多信息。
源分发
构建发行版
bluepyemodel-1.0.24.tar.gz 的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | 0524967c03c34370bd20ae1e41648cf16df33b50334ae5fdb471d654034d8828 |
|
| MD5 | 1018f942d6c76a23c163379115983782 |
|
| BLAKE2b-256 | 6a6ed4438d213fc6b5addbdf50c3773a4dd57d8a43dc889eebcf929daa8d4d7f |
bluepyemodel-1.0.24-py3-none-any.whl 的哈希值
| 算法 | 哈希摘要 | |
|---|---|---|
| SHA256 | d1be4500968efbbfd471660e2b9e31aa2b0059d252e70900a217eb03c57cc589 |
|
| MD5 | dd546585a3a0963c594ec9ff1f948580 |
|
| BLAKE2b-256 | c1ff294023cdd2973f83b7793e40acf18ece77b56ef98592189f8b97553aaf22 |











